PROG0484 - Demultiplexing reads
In bioinformatics, FASTA format is a text-based format for representing either nucleotide sequences or peptide sequences, in which nucleotides or amino acids are represented using single-letter codes. The format originates from the FASTA software package, but has now become a de facto standard in the field of bioinformatics. FASTA files may contain multiple records, where each record corresponds to a single nucleotide or amino acid sequence. A record in FASTA format begins with a single-line description, followed by lines of sequence data. The description line is distinguished from the sequence data by a greater-than (>) symbol in the first column.
The example FASTA file given below contains four records. In order to point out that the number of sequence characters per line is not fixed, we have split the sequences in the example over several lines with a different number of characters for each of the records. But even within a single record it is allowed to have sequence fragments of variable length.
>read1 AAAAAAAAGGTGCGGAGGAAAGCAACACATTTGTTCCTCAGGACTCTTCA GCGGGAGATATCTGCAGAACCAAACACGCTCAAAGACCCGCGCAAATCGG CAAATTGCCTGACGTAGAACACCGACCTAGCGTGTTTATTATGATACTCG GCACCTCTGACTTAATCAAACGTTGTCGAGGTGGAGATGGTATCATCTGG CGTTAGGCATAACGAGCGTGACACTAGCTTC >read2 CTCTCGGAAGTTTGTCCGCACCGACATAAATAGACTGATACTGATCAGGGGGACGGTACG ACCCACTCTGTCGATCGAACCAGTGACCTGTTCGCTTCGTAACTGGCCAGACGATAGATC TTAGCATAACCGACGCGAAGTGTGGCAGATAAGATCCCAAGGTAGTAAATAGTACATATT AGTGGTCAACAGGTTTTTAGCGCAGCAGCTGATCTATGCAATTGACTGCAACACCATGAC GTAGGTTGCTTCTATAAGAACAAGTTTACCGCTACGAATTCGCGTCGGTTCCGTACATA >read3 AAAAAAAAACGTCAGAAACGTGAGTAGGGTTACCCACACGCATCTAAGAATGCTCGGGCAATGTGACGGT ATGAAGTTGAGAGCCTCTGTTACCCTCCACCTGATGGGGTGGCGGCCATTTACAGTATTGCTTAGCGCAC TCAGATATACGCATGATGGGACTGATTCCCCAGGCGAGTACGTAGTCACCCGCGGCGACTCGACAAGGAT ACTATTATCAGGGTTCTCCCCGGGAGGAGGTATTAAGA >read4 AAAAAAAAAATGTCATGACCCTAGAAGGCCCTGCATATACATGGCAGGGCAGTCTATCAGCGCCCATCATCATCGCTGAC GTAGTTGGAGCCGTATCTGTACTGGATCTAGGGGGCATCGTGAACTAGCGAGGTCGTGTGACGCGCTACAAAGGCTCGGC CCATCTGGAACGTCAGACGAGGCTTCTCTACGGGGGTCCTGCCGTGGGCTATTGAGGTGCAAAGTTCGAATTCGGCACTG TCGCGTGTAATTGAATTCGTGCCCAGT
Determining an organism's complete genome (called genome sequencing) forms a central task of bioinformatics. Unfortunately, we still don't possess the microscope technology to zoom into the nucleotide level and determine the sequence of a genome's nucleotides, one at a time. However, researchers can apply chemical methods to generate and identify much smaller snippets of DNA, called reads.
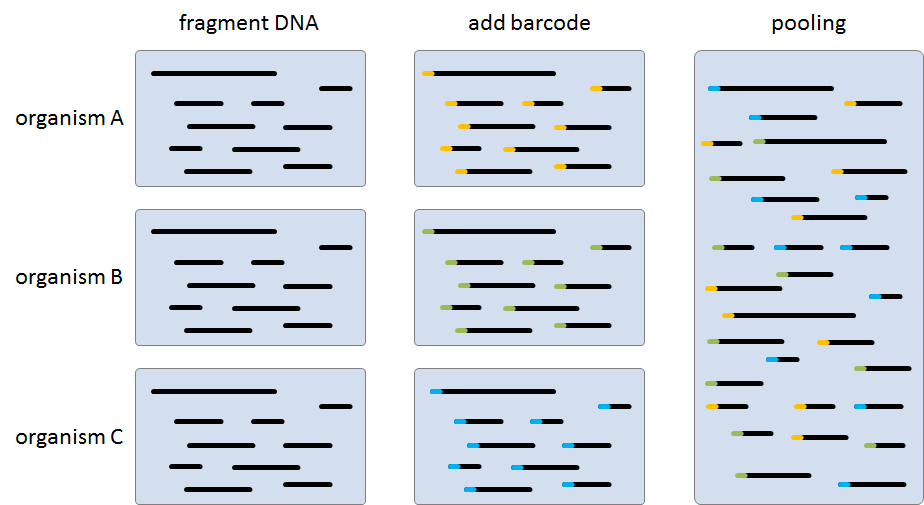
Because the current generation of sequencing machines produce so many reads at once, it is possible to sequence different organisms in parallel. This is done by fragmenting the DNA of the organisms, and sequencing these fragments in a single run. In order to link the sequenced fragments to the organism they originate from, each of the fragments is labeled with some kind of an organism-specific barcode. 454 Life Sciences (Roche) sequencers use a barcode of eight nucleotides that prefix each of the sequence fragments.
Assignment
You are given a FASTA file containing the reads generated by a single multiplexed run on a 454 Life Sciences sequencer. Your task is to split this file into multiple FASTA files, each containing the reads grouped per label. This is done in the following way:
- Implement a function outputFasta that can be used to output a single sequence in FASTA format. The function takes two string arguments: a description and the sequence itself. In generating the output, the function must fragment the sequence into fixed-length strings (except for the last fragment). The default length is 80 characters, but specific lengths can be passed to the optional argument width. By default, the function prints out the sequence in FASTA format. However, if a file object that is opened for writing is passed to the optional argument file, the function instead must write the FASTA formatted sequence to this file. Make sure that each line written by the function, including the last line, ends with a newline ('\n').
- Use the function outputFasta to implement a function demultiplexFasta. The function demultiplexFasta takes as its argument the location of a FASTA file containing a series of reads generated by a single multiplexed run on a 454 Life Sciences sequencer. The function must write all FASTA records from the given file that have the label XXXXXXXX to the file XXXXXXXX.fasta (existing files must be overwritten). In doing so, the order of the records in the given FASTA file must be retained. This procedure must be executed for all labels that prefix the reads in the given file. In writing FASTA records to the new files, the function must remove the labels from the sequences and must split the sequences into fixed-length fragments. The default length is 80 characters, but specific lengths can be passed to the optional argument width.
Example
In the following interactive session we assume the current directory to contain the text file reads.fasta. The content of this file is the same as the example FASTA file displayed in the introduction.
>>> outputFasta('read1', 'GGTGCGGAGGAAAGCAACACATTTGT', width=10) >read1 GGTGCGGAGG AAAGCAACAC ATTTGT >>> output = open('out.fasta', 'w') >>> outputFasta('read2', 'AGTTTGTCCGCACCGACATAAATAGA', width=10, file=output) >>> outputFasta('read3', 'ACGTCAGAAACGTGAG', width=10, file=output) >>> output.close() >>> print(open('out.fasta', 'r').read(), end='') >read2 AGTTTGTCCG CACCGACATA AATAGA >read3 ACGTCAGAAA CGTGAG >>> demultiplexFasta('reads.fasta', width=60) >>> print(open('AAAAAAAA.fasta', 'r').read(), end='') >read1 GGTGCGGAGGAAAGCAACACATTTGTTCCTCAGGACTCTTCAGCGGGAGATATCTGCAGA ACCAAACACGCTCAAAGACCCGCGCAAATCGGCAAATTGCCTGACGTAGAACACCGACCT AGCGTGTTTATTATGATACTCGGCACCTCTGACTTAATCAAACGTTGTCGAGGTGGAGAT GGTATCATCTGGCGTTAGGCATAACGAGCGTGACACTAGCTTC >read3 ACGTCAGAAACGTGAGTAGGGTTACCCACACGCATCTAAGAATGCTCGGGCAATGTGACG GTATGAAGTTGAGAGCCTCTGTTACCCTCCACCTGATGGGGTGGCGGCCATTTACAGTAT TGCTTAGCGCACTCAGATATACGCATGATGGGACTGATTCCCCAGGCGAGTACGTAGTCA CCCGCGGCGACTCGACAAGGATACTATTATCAGGGTTCTCCCCGGGAGGAGGTATTAAGA >read4 AATGTCATGACCCTAGAAGGCCCTGCATATACATGGCAGGGCAGTCTATCAGCGCCCATC ATCATCGCTGACGTAGTTGGAGCCGTATCTGTACTGGATCTAGGGGGCATCGTGAACTAG CGAGGTCGTGTGACGCGCTACAAAGGCTCGGCCCATCTGGAACGTCAGACGAGGCTTCTC TACGGGGGTCCTGCCGTGGGCTATTGAGGTGCAAAGTTCGAATTCGGCACTGTCGCGTGT AATTGAATTCGTGCCCAGT >>> print(open('CTCTCGGA.fasta', 'r').read(), end='') >read2 AGTTTGTCCGCACCGACATAAATAGACTGATACTGATCAGGGGGACGGTACGACCCACTC TGTCGATCGAACCAGTGACCTGTTCGCTTCGTAACTGGCCAGACGATAGATCTTAGCATA ACCGACGCGAAGTGTGGCAGATAAGATCCCAAGGTAGTAAATAGTACATATTAGTGGTCA ACAGGTTTTTAGCGCAGCAGCTGATCTATGCAATTGACTGCAACACCATGACGTAGGTTG CTTCTATAAGAACAAGTTTACCGCTACGAATTCGCGTCGGTTCCGTACATA
In de bioinformatica worden DNA sequenties vaak opgeslaan in tekstbestanden die ingedeeld worden volgens het FASTA formaat. De sequenties zelf worden voorgesteld als strings die enkel bestaan uit hoofdletters of kleine letters. FASTA bestanden kunnen meerdere records bevatten, waarbij elke record één enkele DNA sequentie voorstelt. Een record bestaat uit één regel met een beschrijving, gevolgd door één of meer regels met de opeenvolgende fragmenten van de sequentie. Regels met een beschrijving kunnen onderscheiden worden van regels met een sequentiefragment door het feit dat het eerste karakter van de beschrijvingsregels een groter-dan teken (>) is.
Het FASTA bestand in onderstaand voorbeeld bevat vier records. Om aan te geven dat het aantal sequentiekarakters per regel niet vast ligt, hebben we de sequenties in het voorbeeld telkens opgesplitst over verschillende regels die een vast aantal karakters bevatten. Maar zelfs binnen één record is het toegelaten om te werken met sequentiefragmenten van variabele lengte.
>read1 AAAAAAAAGGTGCGGAGGAAAGCAACACATTTGTTCCTCAGGACTCTTCA GCGGGAGATATCTGCAGAACCAAACACGCTCAAAGACCCGCGCAAATCGG CAAATTGCCTGACGTAGAACACCGACCTAGCGTGTTTATTATGATACTCG GCACCTCTGACTTAATCAAACGTTGTCGAGGTGGAGATGGTATCATCTGG CGTTAGGCATAACGAGCGTGACACTAGCTTC >read2 CTCTCGGAAGTTTGTCCGCACCGACATAAATAGACTGATACTGATCAGGGGGACGGTACG ACCCACTCTGTCGATCGAACCAGTGACCTGTTCGCTTCGTAACTGGCCAGACGATAGATC TTAGCATAACCGACGCGAAGTGTGGCAGATAAGATCCCAAGGTAGTAAATAGTACATATT AGTGGTCAACAGGTTTTTAGCGCAGCAGCTGATCTATGCAATTGACTGCAACACCATGAC GTAGGTTGCTTCTATAAGAACAAGTTTACCGCTACGAATTCGCGTCGGTTCCGTACATA >read3 AAAAAAAAACGTCAGAAACGTGAGTAGGGTTACCCACACGCATCTAAGAATGCTCGGGCAATGTGACGGT ATGAAGTTGAGAGCCTCTGTTACCCTCCACCTGATGGGGTGGCGGCCATTTACAGTATTGCTTAGCGCAC TCAGATATACGCATGATGGGACTGATTCCCCAGGCGAGTACGTAGTCACCCGCGGCGACTCGACAAGGAT ACTATTATCAGGGTTCTCCCCGGGAGGAGGTATTAAGA >read4 AAAAAAAAAATGTCATGACCCTAGAAGGCCCTGCATATACATGGCAGGGCAGTCTATCAGCGCCCATCATCATCGCTGAC GTAGTTGGAGCCGTATCTGTACTGGATCTAGGGGGCATCGTGAACTAGCGAGGTCGTGTGACGCGCTACAAAGGCTCGGC CCATCTGGAACGTCAGACGAGGCTTCTCTACGGGGGTCCTGCCGTGGGCTATTGAGGTGCAAAGTTCGAATTCGGCACTG TCGCGTGTAATTGAATTCGTGCCCAGT
Het bepalen van het volledige genoom van een organisme (genoomsequenering) is één van de hoekstenen van de bioinformatica. Helaas beschikken we nog steeds niet over microscopische technologie die toelaat om tot op nucleotideniveau in te zoomen en zo de reeks nucleotiden van een genoom één voor één af te lezen. Onderzoekers beschikken echter wel over chemische methoden die kunnen toegepast worden voor het genereren en identificeren van kortere DNA fragmenten. In deze context worden de korte fragmenten reads genoemd.
Omdat de huidige generatie sequeneringstoestellen zoveel reads in één keer kunnen genereren, is het mogelijk om verschillende organismen tegelijkertijd te sequeneren. Hierbij wordt het DNA van de organismen in fragmenten geknipt, en worden deze fragmenten samen gesequeneerd in één enkele run. Om achteraf de afzonderlijke reads te kunnen koppelen aan de individuele organismen, worden de fragmenten per organisme gelabeld met een soort barcode. Bij sequeneringstoestellen van 454 Life Sciences (Roche) bestaat deze barcode uit acht nucleotiden die aan het begin van elk fragment toegevoegd worden.
Opgave
Gegeven is een FASTA bestand dat de reads bevat die gegenereerd werden door een run met een 454 Life Sciences sequeneringstoestel. Gevraagd wordt om dit bestand op te splitsen in verschillende FASTA bestanden, die de reads groeperen per label. Hiervoor ga je als volgt te werk:
- Schrijf een functie toonFasta waarmee één enkele sequentie kan uitgeschreven worden in FASTA formaat. Aan de functie moeten een beschrijving en een sequentie doorgegeven worden. Bij het uitschrijven moet de sequentie opgedeeld worden in fragmenten met een vaste breedte (behalve het laatste fragment). De standaardbreedte is 80 karakters, maar deze kan ook worden ingesteld aan de hand van het optionele argument breedte. Standaard moet de functie de sequentie in FASTA formaat uitschrijven. Als aan het optionele argument bestand echter een bestandsobject wordt doorgegeven dat geopend is om te schrijven, dan moet de functie de sequentie in FASTA formaat wegschrijven naar dit bestand. Zorg ervoor dat elke regel die door het bestand wordt uitgeschreven, ook de laatste, eindigt op een newline ('\n').
- Gebruik de functie toonFasta om een functie demultiplexFasta te schrijven. Aan de functie demultiplexFasta moet de locatie van een FASTA bestand doorgegeven worden, dat een reeks reads bevat die gegenereerd werden door een run met een 454 Life Sciences sequeneringstoestel. De functie moet alle FASTA records in het gegeven bestand met label XXXXXXXX wegschrijven naar het bestand XXXXXXXX.fasta (bestaande bestanden moeten hierbij overschreven worden). Hierbij moet de volgorde van de records uit het gegeven FASTA bestand aangehouden worden. Deze procedure moet uitgevoerd worden voor alle labels die in de reads van het gegeven bestand voorkomen. Bij het wegschrijven van de FASTA records naar de nieuwe bestanden moet de functie het label uit de sequenties verwijderen en moeten de sequenties opgedeeld worden in fragmenten van een vast aantal karakters. De standaardbreedte is 80 karakters, maar deze kan ook worden ingesteld aan de hand van het optionele argument breedte.
Voorbeeld
Bij onderstaande voorbeeldsessie gaan we ervan uit dat het bestand reads.fasta zich in de huidige directory bevindt. De inhoud van dit bestand is dezelfde als het voorbeeld FASTA bestand dat we in de inleiding hebben weergegeven.
>>> toonFasta('read1', 'GGTGCGGAGGAAAGCAACACATTTGT', breedte=10) >read1 GGTGCGGAGG AAAGCAACAC ATTTGT >>> uitvoer = open('out.fasta', 'w') >>> toonFasta('read2', 'AGTTTGTCCGCACCGACATAAATAGA', breedte=10, bestand=uitvoer) >>> toonFasta('read3', 'ACGTCAGAAACGTGAG', breedte=10, bestand=uitvoer) >>> uitvoer.close() >>> print(open('out.fasta', 'r').read(), end='') >read2 AGTTTGTCCG CACCGACATA AATAGA >read3 ACGTCAGAAA CGTGAG >>> demultiplexFasta('reads.fasta', breedte=60) >>> print(open('AAAAAAAA.fasta', 'r').read(), end='') >read1 GGTGCGGAGGAAAGCAACACATTTGTTCCTCAGGACTCTTCAGCGGGAGATATCTGCAGA ACCAAACACGCTCAAAGACCCGCGCAAATCGGCAAATTGCCTGACGTAGAACACCGACCT AGCGTGTTTATTATGATACTCGGCACCTCTGACTTAATCAAACGTTGTCGAGGTGGAGAT GGTATCATCTGGCGTTAGGCATAACGAGCGTGACACTAGCTTC >read3 ACGTCAGAAACGTGAGTAGGGTTACCCACACGCATCTAAGAATGCTCGGGCAATGTGACG GTATGAAGTTGAGAGCCTCTGTTACCCTCCACCTGATGGGGTGGCGGCCATTTACAGTAT TGCTTAGCGCACTCAGATATACGCATGATGGGACTGATTCCCCAGGCGAGTACGTAGTCA CCCGCGGCGACTCGACAAGGATACTATTATCAGGGTTCTCCCCGGGAGGAGGTATTAAGA >read4 AATGTCATGACCCTAGAAGGCCCTGCATATACATGGCAGGGCAGTCTATCAGCGCCCATC ATCATCGCTGACGTAGTTGGAGCCGTATCTGTACTGGATCTAGGGGGCATCGTGAACTAG CGAGGTCGTGTGACGCGCTACAAAGGCTCGGCCCATCTGGAACGTCAGACGAGGCTTCTC TACGGGGGTCCTGCCGTGGGCTATTGAGGTGCAAAGTTCGAATTCGGCACTGTCGCGTGT AATTGAATTCGTGCCCAGT >>> print(open('CTCTCGGA.fasta', 'r').read(), end='') >read2 AGTTTGTCCGCACCGACATAAATAGACTGATACTGATCAGGGGGACGGTACGACCCACTC TGTCGATCGAACCAGTGACCTGTTCGCTTCGTAACTGGCCAGACGATAGATCTTAGCATA ACCGACGCGAAGTGTGGCAGATAAGATCCCAAGGTAGTAAATAGTACATATTAGTGGTCA ACAGGTTTTTAGCGCAGCAGCTGATCTATGCAATTGACTGCAACACCATGACGTAGGTTG CTTCTATAAGAACAAGTTTACCGCTACGAATTCGCGTCGGTTCCGTACATA
| Added by: | Peter Dawyndt |
| Date: | 2014-08-17 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |


 RSS
RSS