PROG0316 - Primordial soup
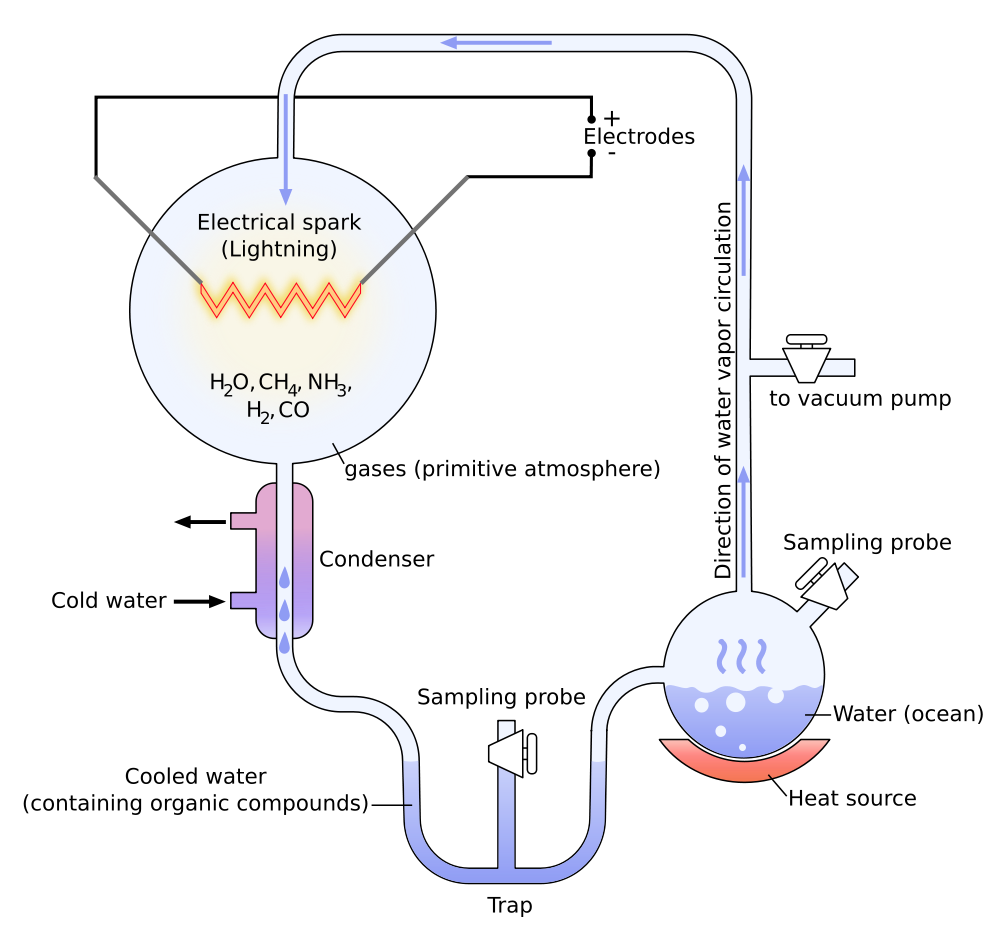
The Miller-Urey experiment is a famous experiment that was conducted in 1953 by Stanley Miller and Harold Urey at the University of Chicago (now the University of California), San Diego and published the following year. The experiment used water, methane, ammonia and hydrogen. The chemicals were sealed inside a sterile array of glass flasks connected in a loop, with one flask half-full of liquid water and another flask containing a pair of electrodes. The liquid water was heated to induce evaporation, sparks were fired between the electrodes to simulate lightning through the atmosphere and water vapor, and then the atmosphere was cooled again so that the water could condense and trickle back into the first flask in a continuous cycle.
Within a day, the mixture has turned pink in color, and at the end of two weeks of continuous operation, Miller and Urey observed that as much as 10–15% of the carbon within the system was now in the form of organic compounds. Two percent of the carbon had formed amino acids that are used to make proteins in living cells, with glycine as the most abundant. Sugars were also formed. Nucleic acids were not formed within the reaction. 18% of the methane-molecules became bio-molecules. The rest turned into hydrocarbons like bitumen. The experiment was seen by some as a proof of how life on Earth was synthesized from inorganic compounds — the so-called primordial soup. A process called abiogenesis.
Assignment
In this exercise, both molecules and mixtures are represented as dictionaries whose keys are formed by the symbolic names of the atoms that occur in it and the corresponding values indicate the number of contained atoms. You are asked to:
- Write a function abiogenesis that takes a molecule and a mixture as its arguments. The function must return a boolean that indicates whether or not the molecule can be formed by the atoms contained in the mixture. The molecule can be formed if all its atoms are sufficiently contained in the mixture.
- Write a function readMolecules that takes the location of a text file as its argument. Each line of the file contains the name of a molecule, followed by a description of the atomic composition of the molecule. This description is formatted as a listing of the symbolic names of the atoms, each time followed by the number of atoms contained in the molecule. All fields in this list are separated by one or more spaces. The function must return a dictionary, whose keys are the names of the molecules that occur in the file. Each molecule name is mapped to a dictionary that describes the atomic composition of the molecule.
- Write a function experiment1 that takes two dictionaries as its arguments. The first dictionary contains the composition of a series of molecules that are targeted as the end product of the experiment, and has the same format as the dictionary returned by the function readMolecules. The second dictionary describes the mixture used in the experiment. The function must return an alphabetically ordered list of the molecule names from the first dictionary that can be formed based on the given mixture. Note that each molecule needs to be treated in isolation: there is no need to check whether there are sufficient atoms in the mixture to form all the molecules from the first dictionary.
- Write a function experiment2 that takes the same two dictionary arguments as the function experiment1. The function must return a Boolean value that indicates whether or not there are sufficient atoms in the mixture to form one molecule for each molecule contained in the first dictionary.
Example
In the following interactive session we assume that the text file molecules.txt is located in the current directory.
>>> soup = {'N':2, 'Na':2, 'O':5, 'H':3} >>> abiogenesis({'Si': 1, 'O': 2}, soup) False >>> abiogenesis({'H': 3, 'N': 1}, soup) True >>> molecules = readMolecules('molecules.txt') >>> molecules['sand'] {'Si': 1, 'O': 2} >>> molecules['ammonia'] {'H': 3, 'N': 1} >>> experiment1(molecules, soup) ['ammonia', 'lye'] >>> molecules1 = {'ammonia': {'H': 3, 'N': 1}, 'lye': {'Na': 1, 'O': 1, 'H': 1}} >>> experiment2(molecules1, soup) True >>> molecules = {'sand': {'Si': 1, 'O': 2}, 'ammonia': {'H': 3, 'N': 1}} >>> experiment2(molecules, soup) False
References
Het Miller-Urey experiment is een beroemd geworden experiment dat in 1952 aan de Universiteit van Chicago werd uitgevoerd door Stanley Miller en Harold Urey. In het experiment werd een mengsel van methaan, ammoniak, waterdamp en diwaterstof verwarmd en blootgesteld aan elektrische ladingen. De samenstelling van het mengsel werd gekozen op grond van wat men op dat moment aannam over de atmosfeer van voor er leven was op de jonge Aarde. De elektrische ladingen moesten de bliksem simuleren.
Na een week continu te hebben gedraaid met hun opstelling vonden Miller en Urey dat 10% tot 15% van de koolstof in het systeem zich nu in organische verbindingen bevond. Twee procent had aminozuren gevormd, waaronder 13 van de 20 die tegenwoordig door levende cellen gebruikt worden. Het aminozuur dat het meest voorkwam was glycine. Er werden ook suikers en lipiden gevormd. De bouwstenen van DNA en RNA (nucleotiden) ontbraken echter in zijn geheel. Simpele versies van nucleotiden zoals nucleosiden (nucleotiden zonder de fosfaatgroep) werden ook niet gevonden. Het experiment wordt door sommigen gezien als aanwijzing van hoe het leven op Aarde uit anorganische stoffen — de zogenaamde oersoep — is ontstaan. Een proces dat abiogenese genoemd wordt.
Opgave
In deze opgave stellen we een molecule of een mengsel voor als een dictionary, waarvan de sleutels gevormd worden door de symbolische namen van de atomen die erin voorkomen en de corresponderende waarden aangeven hoeveel atomen erin voorkomen. Gevraagd wordt:
- Schrijf een functie abiogenese waaraan een molecule en een mengsel als argument moeten doorgegeven worden. De functie moet een Booleaanse waarde teruggeven die aangeeft of het molecule al dan niet kan gevormd worden met de atomen die voorkomen in het mengsel. Het molecule kan gevormd worden, als alle atomen van het molecule in voldoende mate in het mengsel aanwezig zijn.
- Schrijf een functie leesMoleculen waaraan de locatie van een tekstbestand moet doorgegeven worden. Elke regel van dit bestand bevat de naam van een molecule, gevolgd door een beschrijving van de atomaire samenstelling van de molecule. Deze omschrijving bestaat uit een oplijsting van de symbolische namen van de atomen, telkens gevolgd door het aantal van dat type atomen dat in het molecule voorkomt. Alle velden worden hierbij van elkaar gescheiden door één of meer spaties. De functie moet een dictionary als resultaat teruggeven. De sleutels van deze dictionary worden gevormd door de namen van de moleculen die in het bestand voorkomen. Elke naam van een molecule wordt afgebeeld op een dictionary die de atomaire samenstelling van het molecule beschrijft.
- Schrijf een functie experiment1 waaraan twee dictionaries als argument moeten doorgegeven worden. De eerste dictionary bevat de samenstelling van een aantal moleculen die men op basis van het experiment wil verkrijgen, en heeft hetzelfde formaat als de dictionary die door de functie leesMoleculen wordt teruggegeven. De tweede dictionary beschrijft het mengsel dat bij het experiment gebruikt wordt. De functie moet als resultaat een alfabetisch gerangschikte lijst met de namen van alle moleculen uit de eerste dictionary teruggeven die aan de hand van het gegeven mengsel kunnen gevormd worden. Let wel, elke molecule moet hierbij afzonderlijk behandeld worden: er moet niet nagegaan worden of er voldoende atomen beschikbaar zijn om een reeks moleculen te maken.
- Schrijf een functie experiment2 waaraan dezelfde twee dictionaries als argument moeten doorgegeven worden als bij de functie experiment1. De functie moet een Booleaanse waarde teruggeven die aangeeft of er voldoende atomen in het mengsel aanwezig zijn om van alle moleculen die in het eerste dictionary-argument vervat zitten telkens één molecule te maken.
Voorbeeld
Bij onderstaande voorbeeldsessie gaan we ervan uit dat het tekstbestand moleculen.txt zich in de huidige directory bevindt.
>>> oersoep = {'N':2, 'Na':2, 'O':5, 'H':4} >>> abiogenese({'Si': 1, 'O': 2}, oersoep) False >>> abiogenese({'H': 3, 'N': 1}, oersoep) True >>> moleculen = leesMoleculen('moleculen.txt') >>> moleculen['zand'] {'Si': 1, 'O': 2} >>> moleculen['ammoniak'] {'H': 3, 'N': 1} >>> experiment1(moleculen, oersoep) ['ammoniak', 'natronloog'] >>> moleculen1 = {'ammoniak': {'H': 3, 'N': 1}, 'natronloog': {'Na': 1, 'O': 1, 'H': 1}} >>> experiment2(moleculen1, oersoep) True >>> moleculen2 = {'zand': {'Si': 1, 'O': 2}, 'ammoniak': {'H': 3, 'N': 1}} >>> experiment2(moleculen2, oersoep) False
Bronnen
| Added by: | Peter Dawyndt |
| Date: | 2013-01-28 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS