PROG0248 - Protein mass
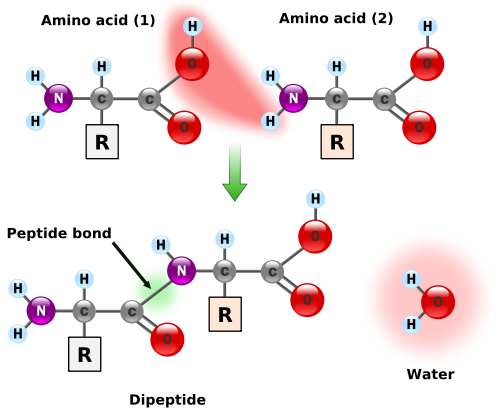
When two amino acids link together, they form a peptide bond, which releases a molecule of water (left figure). Thus, after a series of amino acids have been linked together into a polypeptide, every pair of adjacent amino acids has lost one molecule of water, meaning that a polypeptide containing $n$ amino acids has had $n-1$ water molecules removed.
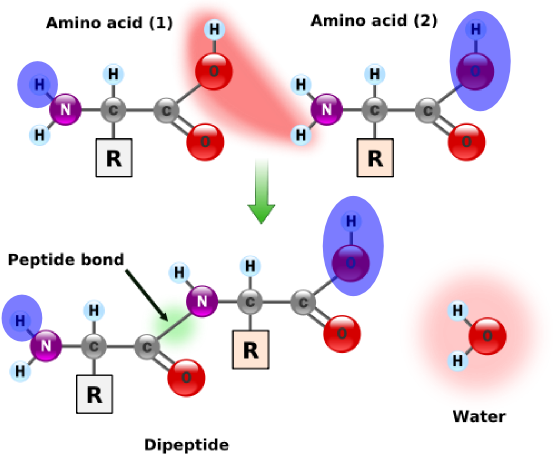
More generally, a residue is a molecule from which a water molecule has been removed. Every amino acid in a protein are residues except the leftmost and the rightmost ones. These outermost amino acids are special in that one has an "unstarted" peptide bond, and the other has an "unfinished" peptide bond. Between them, the two molecules have a single "extra" molecule of water (see the atoms marked in blue in the figure on the right). Thus, the mass of a protein is the sum of masses of all its residues plus the mass of a single water molecule.
There are two standard ways of computing the mass of a residue by summing the masses of its individual atoms. Its monoisotopic mass is computed by using the principal (most abundant) isotope of each atom in the amino acid, whereas its average mass is taken by taking the average mass of each atom in the molecule (over all naturally appearing isotopes).
Many applications in proteomics rely on mass spectrometry, an analytical chemical technique used to determine the mass, elemental composition, and structure of molecules. In mass spectrometry, monoisotopic mass is used more often than average mass, and so all amino acid masses are assumed to be monoisotopic unless otherwise stated.
The standard unit used in mass spectrometry for measuring mass is the atomic mass unit, which is also called the dalton (Da) and is defined as one twelfth of the mass of a neutral atom of carbon-12. The mass of a protein is the sum of the monoisotopic masses of its amino acid residues plus the mass of a single water molecule (whose monoisotopic mass is 18.01056 Da).
In some applications of mass spectrometry, the complication of having to distinguish between residues and non-residues is avoided by only considering peptides excised from the middle of the protein. This is a relatively safe assumption because in practice, peptide analysis is often performed in tandem mass spectrometry. In this special class of mass spectrometry, a protein is first divided into peptides, which are then broken into ions for mass analysis.
Assignment
To calculate the mass of a given protein, you have to implement the following functions:
- A function massTable that takes the location of a text file as its argument. Each line of this text file should contain an uppercase letter, followed by one or more spaces and a real-valued number. All uppercase letters in the file must be distinct, and represent the different amino acids. The read-valued numbers represent the monoisotopic mass of the amino acid on the same line. The function must return a dictionary that maps each amino acid in the given file onto its monoisotopic mass.
- A function proteinMass that takes two arguments: a protein and a mass table. The protein is given as a string that only contains uppercase letters, representing the amino acid sequence of the protein. The mass table is represented as a dictionary that maps each upper case letter that is used to represent an amino acid onto its monoisotopic mass. In addition, the function has an optional parameter peptide that indicates whether or not the given protein string represents a peptide that was excised from the middle of a protein (default value: False). The function must return the mass of the given protein.
Example
In the following interactive session, we assume that the text file mass.txt is located in the current directory.
>>> table = massTable('mass.txt') >>> table['A'] 71.03711 >>> table['E'] 129.04259 >>> table = massTable('mass.txt') >>> proteinMass('SKADYEK', table) 839.40248 >>> proteinMass('SKADYEK', table, peptide=True) 821.3919199999999
Wanneer twee aminozuren aaneenschakelen dan vormen ze een peptidebinding, waarbij één enkele watermolecule wordt vrijgegeven (zie linker figuur). Als bijgevolg een reeks aminozuren aaneengeschakeld zijn tot een polypeptide, dan heeft elk paar aangrenzende aminozuren een watermolecule verloren. Dit betekent dat er uit een polypeptide die bestaat uit $n$ aminozuren in totaal $n - 1$ watermoleculen verwijderd zijn.
Meer algemeen wordt een molecule waaruit een watermolecule verwijderd is een residu genoemd. Alle aminozuren van een eiwit zijn dus residuen, behalve de aminozuren uiterst links en rechts. De buitenste aminozuren zijn speciaal, in die zin dat de ene een "onbegonnen" peptidebinding heeft en de andere een "onafgewerkte" peptidebinding. Samen delen deze twee moleculen één "extra" watermolecule (zie rechter figuur). De massa van een eiwit kan dus berekend worden als de som van de massa's van de residuen, plus de massa van één enkele watermolecule.
Er zijn twee standaardmanieren om de massa van een residu te bereken, waarbij telkens de massa's van de individuele atomen van het residu worden opgeteld. De mono-isotopische massa wordt berekend op basis van het hoofdisotoop (het isotoop dat meest voorkomt) van elk atoom in het aminozuur, terwijl de gemiddelde massa berekend wordt op basis van de gemiddelde massa van elk atoom in de molecule (over alle natuurlijk voorkomende isotopen heen).
In het onderzoeksdomein van de proteomics doen veel toepassingen beroep op massaspectrometrie, een analytische chemische techniek die gebruikt wordt om de massa, de elementaire samenstelling, en de structuur van moleculen te bepalen. Bij massaspectrometrie wordt doorgaans gebruik gemaakt van de mono-isotopische massa eerder dan van de gemiddelde massa, en worden alle aminozuren verondersteld mono-isotopisch te zijn, tenzij dat expliciet anders staat aangegeven.
De standaardeenheid die in de massaspectrometrie gebruikt wordt voor het meten van massa is de atomaire massa-eenheid, ook wel dalton (Da) genoemd. Deze wordt gedefinieerd als één twaalfde van de massa van een neutraal atoom van koolstof-12. De massa van een eiwit is dan de som van de mono-isotopische massa's van de aminozuren plus de massa van één watermolecule (waarvan de mono-isotopische massa 18,01056 Da bedraagt).
In sommige toepassingen van massaspectrometrie wordt de complicatie van het onderscheid tussen residuen en niet-residuen vermeden door enkel rekening te houden met peptiden die ergens uit het midden van een eiwit geknipt werden. Dit is een relatief veilige veronderstelling, omdat peptide-analyse in de praktijk vaak wordt uitgevoerd in tandem massaspectrometrie. In deze speciale klasse van massaspectrometrie wordt een eiwit eerst opgebroken in peptiden, die daarna nog eens opgebroken worden in ionen voorafgaand aan de massa-analyse.
Opgave
Om de massa van een gegeven eiwit te berekenen, ga je als volgt te werk:
- Schrijf een functie massatabel waaraan de locatie van een tekstbestand moet doorgegeven worden. Elke regel van dit tekstbestand moet een hoofdletter bevatten, gevolgd door één of meerdere spaties en een reëel getal. Alle hoofdletters zijn verschillend, en stellen de verschillende aminozuren voor. Het reëel getal stelt telkens de mono-isotopische massa van het aminozuur op dezelfde regel voor. De functie moet een dictionary teruggeven, waarin elk aminozuur wordt afgebeeld op zijn corresponderende mono-isotopische massa.
- Schrijf een functie eiwitmassa waaraan twee argumenten moeten doorgegeven worden: een eiwitstring en een massatabel. De eiwitstring is een string die enkel bestaat uit hoofdletters die gebruikt worden voor de voorstelling van aminozuren. De massatabel wordt voorgesteld als een dictionary die elk van de hoofdletters voor de aminozuren afbeeldt op de corresponderende mono-isotopische massa. Daarnaast heeft de functie ook nog een optionele parameter peptide, die aangeeft of het gegeven eiwit een peptide voorstelt die ergens uit het midden van een eiwit geknipt werd (standaardwaarde: False). De functie moet de massa van het gegeven eiwit als resultaat teruggeven.
Voorbeeld
Bij onderstaande voorbeeldsessie gaan we ervan uit dat het tekstbestand massa.txt zich in de huidige directory bevindt.
>>> tabel = massatabel('massa.txt') >>> tabel['A'] 71.03711 >>> tabel['E'] 129.04259 >>> eiwitmassa('SKADYEK', tabel) 839.40248 >>> eiwitmassa('SKADYEK', tabel, peptide=True) 821.3919199999999
| Added by: | Peter Dawyndt |
| Date: | 2012-05-08 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS