PROG0206 - Levenshtein
The Levenshtein distance between two DNA sequences is the minimal amount of treatments that are necessary in order to convert one sequence to another. This linear measure is named after Vladimir Levenshtein, who invented it in 1965. Apart from its use in the spelling checker, the Levenshtein disctance is also used to make astimates of the evolutionary distance betwee two DNA sequences (where the variants of the Needleman-Wunsh or the Smith-Waterman are usually used). Here, a DNA sequence can be represented by a sequence of symbols that consists only of the letters A, C, G and T. The collection of valid treatments is then insertion (inserting a letter), deletion (deleting a letter) and substitution (replacing a letter by another letter).
For the calculation of the Levenshtein
distance between two given DNA sequences, a two-dimensional matrix
$d(0,\ldots,m;0,\ldots,n)$ is used, where $m$ represents the length of the
first DNA sequence and $n$ the length of the second DNA sequence.
Initially you fill out the first column of the matrix $d$ with the numbers
$0,1,\ldots,m$ and the first row with the numbers $0,1,\ldots,n$. The
other numbers are calculated from left to right and from top to bottom,
where the value on row $i$ and column $j$ are given by
\[
d_{i,j} = \textrm{minimum}
\left\{
\begin{array}{ll}
d_{i-1,j}+1&\textrm{deletion}\\
d_{i,j-1}+1&\textrm{insertion}\\
d_{i-1,j-1}+\textrm{cost}\quad\quad&\textrm{substitution}\\
\end{array}
\right.
\]
Here, the cost is equal to 0 if the $i$th letter of the first DNA sequence
equals the $j$th letter of the second DNA sequence. Otherwise, the cost
equals 1. At the end of this procedure, the Levenshtein distance can be
read as the value on the bottom right of the matrix.
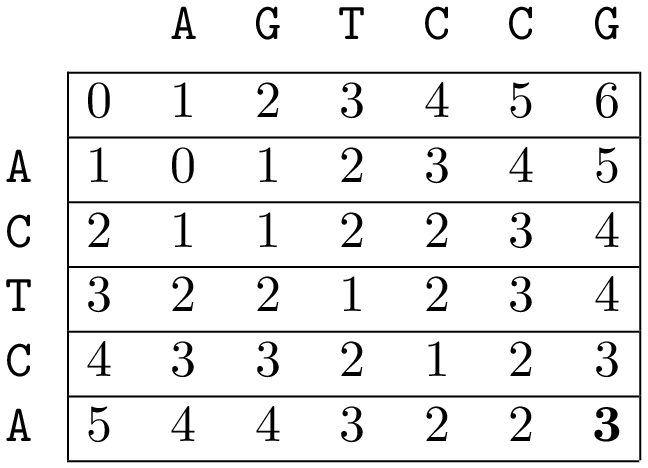
An example, the Levenshtein distance between ACTCA and AGTCCG is 3 and can thus be given:
- ACTCA $\longrightarrow$ AGTCA (first C replaced by G)
- AGTCA $\longrightarrow$ AGTCC (A at the back replaced by C)
- AGTCC $\longrightarrow$ AGTCCG (add G in the back)
The end result after calculating the two-dimensional matrix $d$ looks as follows:
Assignment
Define a class Sequence which can be used to make instances of valid DNA sequences. The objects of the class Sequence must support the following methods:
- The initializing method __init__ allows to make DNA sequence objects based on a given string (that is given as an argument). Such objects have an attribute seq that refers to a string that only consists of the letters A, C, G and T. The given string must be converted by the method __init__ to upper case letters, and the validity of it must be checked by calling the initializing method validate. Study the example below to find out what must happen if the given string does not represent a valid DNA sequence.
- The method validate prints the value True if the value of the attribute seq only consists of the letters A, C, G and T. Otherwise, the value False is printed. To this method, no arguments must be given.
- The method distance prints the Levenshtein distance between the current Sequence object and another Sequence object that must be given to the method as an argument.
Example
>>> seq1 = Sequence('ACTCA') >>> seq2 = Sequence('AGTCCG') >>> seq1.distance(seq2) 3 >>> seq3 = Sequence('GTCNAAC') Traceback (most recent call last): AssertionError: invalid sequence
De Levenshteinafstand tussen twee DNA sequenties is de minimale hoeveelheid bewerkingen die nodig zijn om de ene sequentie om te zetten in de andere. Deze afstandsmaat is genoemd naar Vladimir Levenshtein, die hem in 1965 uitvond. Naast zijn toepassing in de spellingscontrole, wordt de Levenshteinafstand ook gebruikt om schattingen te maken van de evolutionaire afstand tussen twee DNA sequenties (waarbij dan meestal gebruik wordt gemaakt van de varianten van Needleman-Wunsch of Smith-Waterman). Hierbij kan een DNA sequentie worden voorgesteld door een tekenreeks die enkel bestaat uit de lettertekens A, C, G en T. De verzameling geldige bewerkingen is dan insertie (letter invoegen), deletie (letter verwijderen) en substitutie (letter veranderen door een andere letter).
Voor de berekening van de Levenshteinafstand
tussen twee gegeven DNA sequenties wordt gebruik gemaakt van een
tweedimensionale matrix $d(0,\ldots,m;0,\ldots,n)$, waarbij $m$ de lengte
van de eerste DNA sequentie voorstelt en $n$ de lengte van de tweede DNA
sequentie. Initieel vul je de eerste kolom van de matrix $d$ met de
getallen $0,1,\ldots,m$ en de eerste rij met de getallen $0,1,\ldots,n$.
De andere getallen worden van links naar rechts en van boven naar onder
berekend, waarbij de waarde op rij $i$ en kolom $j$ wordt gegeven door
\[
d_{i,j} = \textrm{minimum}
\left\{
\begin{array}{ll}
d_{i-1,j}+1&\textrm{deletie}\\
d_{i,j-1}+1&\textrm{insertie}\\
d_{i-1,j-1}+\textrm{kost}\quad\quad&\textrm{substitutie}\\
\end{array}
\right.
\]
Hierbij is de kost gelijk aan 0 als de $i$-de letter van de eerste DNA
sequentie gelijk is aan de $j$-de letter van de tweede DNA sequentie.
Anders is de kost gelijk aan 1. Op het einde van deze procedure kan de
Levenshteinafstand worden uitgelezen als de waarde rechtsonderaan in de
matrix.
Een voorbeeld, de Levenshteinafstand tussen ACTCA en AGTCCG is 3 en kan als volgt weergegeven worden:
- ACTCA $\longrightarrow$ AGTCA (eerste C vervangen door G)
- AGTCA $\longrightarrow$ AGTCC (A achteraan vervangen door C)
- AGTCC $\longrightarrow$ AGTCCG (achteraan G toevoegen)
Het eindresultaat na berekening van de tweedimensionale matrix $d$ ziet er dan als volgt uit:
Opgave
Definieer een klasse Sequentie waarmee instanties van geldige DNA sequenties kunnen aangemaakt worden. De objecten van de klasse Sequentie moeten ondersteuning bieden aan de volgende methoden:
- De initialisatiemethode __init__ laat toe om DNA sequentieobjecten aan te maken op basis van een gegeven string (die als argument wordt meegegeven). Dergelijke objecten hebben een attribuut seq dat verwijst naar een string die enkel bestaat uit de letters A, C, G en T. De meegeleverde string moet door de methode __init__ omgezet worden naar hoofdletters, en de geldigheid ervan moet gecontroleerd worden door in de initialisatiemethode de methode valideer aan te roepen. Bestudeer onderstaand voorbeeld om te achterhalen wat er moet gebeuren indien de doorgegeven string geen geldige DNA sequentie voorstelt.
- De methode valideer geeft de waarde True terug indien de waarde van het attribuut seq enkel bestaat uit de letters A, C, G en T. Anders wordt de waarde False teruggegeven. Aan deze methode moeten geen argumenten doorgegeven worden.
- De methode afstand geeft de Levenshteinafstand terug tussen het huidige Sequentie object en een ander Sequentie object dat als argument aan de methode moet doorgegeven worden.
Voorbeeld
>>> seq1 = Sequentie('ACTCA') >>> seq2 = Sequentie('AGTCCG') >>> seq1.afstand(seq2) 3 >>> seq3 = Sequentie('GTCNAAC') Traceback (most recent call last): AssertionError: ongeldige sequentie
| Added by: | Peter Dawyndt |
| Date: | 2012-01-12 |
| Time limit: | 5s-10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS