| Submit | All submissions | Best solutions | Back to list |
PROG0526 - Double indemnity |
In February 2013, brothers named Elwin and Yohan were arrested for six rapes in France, but both denied the charges. Deciding which is guilty is a tricky affair — they're identical twins, so the genetic difference between them is very slight. Marseille police chief Emmanual Kiehl said, "It could take thousands of separate tests before we know which one of them may be guilty."
This is only the latest in a series of legal conundrums involving identical twins and DNA evidence. During a jewel heist in Germany in January 2009, thieves left behind a drop of sweat on a latex glove. A crime database showed two hits — identical twins Hassan and Abbas O. (under German law their last name was withheld). Both brothers had criminal records for theft and fraud, but both were released. The court ruled, "From the evidence we have, we can deduce that at least one of the brothers took part in the crime, but it has not been possible to determine which one."
Later that year, identical twins Sathis Raj and Sabarish Raj escaped hanging in Malaysia when a judge ruled it was impossible to determine which was guilty of drug smuggling. "Although one of them must be called to enter a defence, I can't be calling the wrong twin to enter his defence," the judge told the court. "I also can't be sending the wrong person to the gallows."
In 2003, a Missouri woman had sex with identical twins Raymon and Richard Miller within hours of one another. When she became pregnant, both men denied fathering the child. In Missouri a man can be named a legal father only if a paternity test shows a 98 percent or higher probability of a DNA match, but the Miller twins both showed a probability of more than 99.9 percent.
"With identical twins, even if you sequenced their whole genome you wouldn't find difference," forensic scientist Bob Gaensslen told ABC News at the time. More recent research shows that this isn't the case, but teasing out the difference can be expensive — in the Marseilles case, police were told that such a test would cost €996,000.
It goes on. In August 2013, British authorities were trying to decide how to prosecute a rape when DNA evidence identified both Mohammed and Aftab Asghar. "It is an unusual case," said prosecutor Sandra Beck. "They are identical twins. The allegation is one of rape. There is further work due."
Assignment
Most differences found in the genomes of identical twins are due to copy-number variations (CNV). These structural variations alter the DNA of a genome such that cells have an abnormal or — for certain genes — a normal variation in the number of copies of one or more sections of the DNA. CNVs correspond to relatively large regions of the genome that have been removed (deletions) or duplicated (insertions) on certain chromosomes. For example, the chromosome that normally has sections in order as A-B-C-D might instead have sections A-B-C-C-C-D (a duplication of C) or A-B-D (a deletion of C).
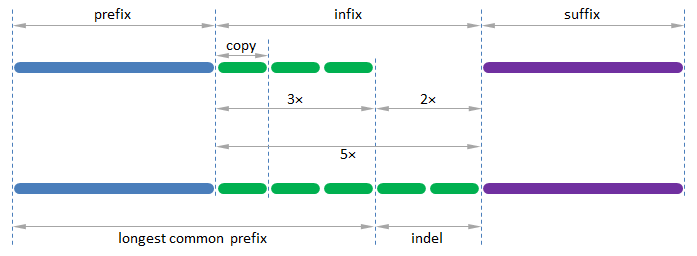
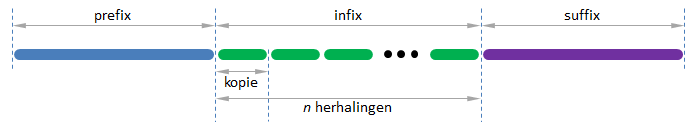
In order to detect CNVs, we assume a DNA sequence in composed as the concatenation of a prefix, followed by an infix and a suffix, where the infix is composed of $n \in \mathbb{N}$ repeats of a DNA-fragment that we call the copy.
In this assignment, we represent a DNA sequence as a string that only contains the uppercase letters A, C, G and T. Now, say that we have two DNA sequences that only differ in the number of repeats of the copy. Based on a comparison of both sequences, we can identify their different components. In order to do so, you proceed as follows:
- Write a function replicate that has one mandatory parameter copy and three optional parameters number (default value: 1), prefix (default value: the empty string) and suffix (default value: the empty string). An positive integer must be passed to the the parameter number, and a DNA sequence must be passed to the other parameters. The function must return the DNA sequence that if composed as the concatenation of the given prefix, followed by an infix and the given suffix, where the infix is composed of the given number of repeats of the given copy.
- Write a function copy_number that takes a DNA sequence $s$. The function must return a tuple that contains a DNA sequence $c$ and an integer $n \in \mathbb{N}_0$, where $c$ is the shortest possible DNA sequence for which the given DNA sequence $s$ is composed out of $n$ repeats of $k$.
- Write a function LGE that takes two DNA sequences. The function also has an optional parameter suffix (default value: False) that takes a Boolean value. If the value False is passed to the parameter suffix, the function must return the longest common prefix (longest common string at the start of the given sequences). If the value True is passed to the parameter suffix, the function must return the longest common suffix (longest common string at the end of the given sequences).
- Now, use the previous two functions to write a function CNV
that takes two DNA sequences. In case both DNA sequences have the same
prefix and suffix, and only differ in the number of intermediate repeats
of the same copy, the function must return a tuple that contains the
(shortest possible) copy and the number of repeats of this copy in the
first and in the second sequence. Otherwise, the function must raise an
AssertionError with the
message no CNV found. Use
the following procedure to identify the different components in the two
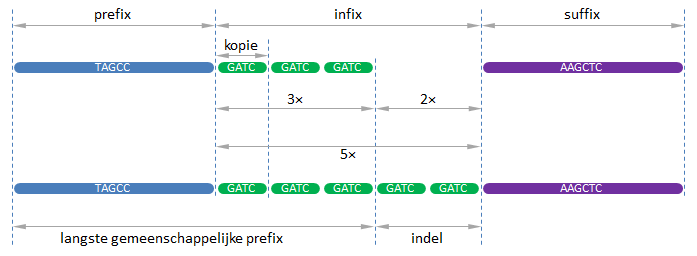
given DNA sequences (see figure below):
- check that the sequences differ, otherwise no CNV is found
- determine the longest common prefix (LCP) of the sequences
- determine the suffix as the remaining part of the shortest sequence (tail not belonging to the LCP)
- check that the longest sequence ends with the suffix found, otherwise no CNV is found
- determine the indel as the part in between the LCP and the suffix of the longest sequence
- determine the shortest possible copy and the number of repeats of that copy from which the indel is composed (in case of CNV the indel is composed of one or more repeats of the copy)
- determine the number of extra repeats of the copy at the end of the LCP
Example
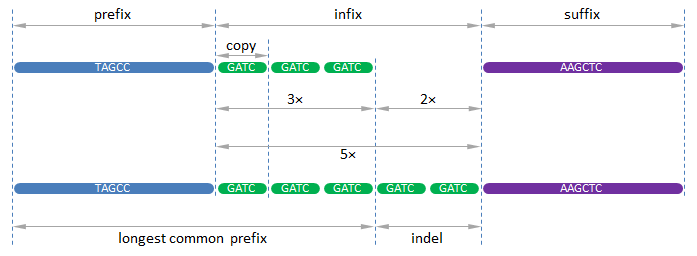
>>> replicate('GATC') 'GATC' >>> replicate('GATC', number=4) 'GATCGATCGATCGATC' >>> replicate('GATC', number=2, prefix='TAGCC') 'TAGCCGATCGATC' >>> replicate(copy='GATC', number=3, suffix='AAGCTC') 'GATCGATCGATCAAGCTC' >>> replicate(copy='GATC', number=3, prefix='TAGCC', suffix='AAGCTC') 'TAGCCGATCGATCGATCAAGCTC' >>> replicate(copy='GATC', number=5, suffix='AAGCTC', prefix='TAGCC') 'TAGCCGATCGATCGATCGATCGATCAAGCTC' >>> copy_number('CTCTCTCTCTCTCTCTCTCTCTCT') # replicate(copy='CT', number=12) ('CT', 12) >>> copy_number('GATCGATCGATCGATC') # replicate(copy='GATC', number=4) ('GATC', 4) >>> copy_number(replicate('GATCGATC', number=2)) ('GATC', 4) >>> copy_number('GATCGATCGATCGATCG') # replicate(copy='GATC', number=4) + 'G' ('GATCGATCGATCGATCG', 1) >>> seq1 = 'TAGCCGATCGATCGATCAAGCTC' # replicate(copy='GATC', number=3, prefix='TAGCC', suffix='AAGCTC') >>> seq2 = 'TAGCCGATCGATCGATCGATCGATCAAGCTC' # replicate(copy='GATC', number=5, suffix='AAGCTC', prefix='TAGCC') >>> LCE(seq1, seq2) 'TAGCCGATCGATCGATC' >>> LCE(seq1, seq2, suffix=True) 'CGATCGATCGATCAAGCTC' >>> LCE(seq1, seq1) 'TAGCCGATCGATCGATCAAGCTC' >>> seq1 = 'TAGCCGATCGATCGATCAAGCTC' # replicate(copy='GATC', number=3, prefix='TAGCC', suffix='AAGCTC') >>> seq2 = 'TAGCCGATCGATCGATCGATCGATCAAGCTC' # replicate(copy='GATC', number=5, suffix='AAGCTC', prefix='TAGCC') >>> CNV(seq1, seq2) ('GATC', 3, 5) >>> CNV(seq2, seq1) ('GATC', 5, 3) >>> CNV(seq1, seq1) Traceback (most recent call last): AssertionError: no CNV found >>> seq3 = 'TAGCGATCGATCGATCGATCGATCAAGCTC' # replicate(copy='GATC', number=5, prefix='TAGC', suffix='AAGCTC') >>> CNV(seq1, seq3) ('GATCGAT', 0, 1) >>> seq4 = 'TAGCCGATCGATCGATCGATCGATCAGCTC' # replicate(copy='GATC', number=5, suffix='AGCTC', prefix='TAGCC') >>> CNV(seq1, seq4) Traceback (most recent call last): AssertionError: no CNV found
In februari 2013 werden de broers Elwin en Yohan in Frankrijk gearresteerd voor een reeks van zes verkrachtingen. Beide ontkennen ze echter alle beschuldigingen. Uitmaken wie van de twee schuldig is, is echter niet zo eenvoudig — het zijn immers identieke tweelingen, waardoor de genetische verschillen tussen de twee miniem zijn. Politiechef Emmanual Kiehl van Marseille zei hierover: "Er zullen waarschijnlijk duizenden verschillende testen nodig zijn voor we kunnen beslissen wie van de twee de dader is."
Deze zaak is de zoveelste in een reeks juridische verwikkelingen waarbij DNA-materiaal wordt aangewend als bewijslast tegen identieke tweelingen. Tijdens een juwelenroof in Duitsland in januari 2009 lieten de dieven een druppel zweet achter op een latex handschoen. In een databank van misdadigers werden twee hits gevonden — identieke tweelingen Hassan en Abbas O. (de Duitse wet vereist geheimhouding van de familienaam van verdachten). Beide broers hebben een strafblad voor diefstal en fraude, maar toch werden ze alletwee vrijgelaten. De rechtbank oordeelde immers: "Uit de bewijslast die voorhanden is, kunnen we afleiden dat ten minste één van de twee broers bij de misdaad betrokken was, maar het was niet mogelijk om uit te maken wie van de twee."
Later dat jaar ontsnapte de identieke tweeling Sathis Raj en Sabarish Raj in Maleisië aan dood door ophanging, toen een rechter oordeelde dat het onmogelijk was om te bepalen wie van de twee schuldig was aan drugssmokkel. "Hoewel één van de twee zou moeten opgeroepen worden om zich te komen verdedigen, kan ik het niet maken om de verkeerde op te roepen", oordeelde de rechter. "Ik kan het ook niet maken om de verkeerde persoon naar de galg te sturen."
In 2003 had een vrouw uit Missouri binnen het tijdsbestek van enkele uren afzonderlijk sex met de identieke tweeling Raymon and Richard Miller. Toen ze zwanger bleek te zijn, ontkenden beide mannen de vader te zijn van het kind. In Missouri wordt een man enkel als de wettelijke vader erkend als een vaderschapstest een kans aangeeft van 98 procent of meer op een DNA-match. In dit geval toonde die voor de Miller-tweeling echter tweemaal een kans van meer dan 99.9 procent.
"Bij identieke tweelingen zou je zelfs geen verschil vinden als je hun volledige genoom zou sequeneren", vertelde de forensische wetenschapper Bob Gaensslen toentertijd aan ABC News. Recenter onderzoek toont echter aan dat dit niet het geval is, maar het uitpluizen van de verschillen kan wel een dure aangelegenheid zijn — in de zaak uit Marseille werd aan de politie verteld dat een dergelijke test €996,000 zou kosten.
En de reeks gaat verder. In Augustus 2013 probeerden de Briste autoriteiten uit te maken hoe ze iemand zouden kunnen vervolgen voor een verkrachting waarbij DNA-materiaal zowel Mohammed als Aftab Asghar had geidentificeerd. "Het is een ongewone zaak", zei officier van justitie Sandra Beck. "Het zijn identieke tweelingen. De beschuldiging is er één van verkrachting, maar er is bijkomend onderzoek nodig."
Opgave
De grootste verschillen in het genoom van identieke tweelingen zijn toe te schrijven aan copy-number variaties (CNV). Bij deze structurele variaties wordt het DNA van een genoom gewijzigd zodat cellen een abnormale of — voor bepaalde genen — een normale variatie hebben in het aantal herhalingen van één of meer DNA-fragmenten. CNVs corresponderen met relatief grote fragmenten van het genoom die verwijderd (deletie) of gedupliceerd (insertie) werden op bepaalde chromosomen. Zo kan een chromosoom dat normaal de fragmenten A-B-C-D heeft, in plaats daarvan de fragmenten A-B-C-C-C-D (een duplicatie van C) of A-B-D (een deletie van C) hebben.
Om CNVs op te sporen, veronderstellen we dat een DNA-sequentie is opgebouwd uit een prefix, gevolgd door een infix en een suffix, waarbij de infix bestaat uit $n \in \mathbb{N}$ herhalingen van een DNA-fragment dat we de kopie noemen.
In deze opgave stellen we DNA-sequenties voor als strings die enkel bestaan uit de hoofdletters A, C, G en T. Veronderstel nu dat we beschikken over twee DNA-sequenties die enkel verschillen in het aantal herhalingen van de kopie. Dan kunnen we op basis van een vergelijking tussen de twee sequenties, de verschillende onderdelen identificeren waaruit ze zijn opgebouwd. Hiervoor gaan we als volgt te werk:
- Schrijf een functie repliceer met één verplichte parameter kopie en drie optionele parameters aantal (standaardwaarde: 1), prefix (standaardwaarde: de lege string) en suffix (standaardwaarde: de lege string). Aan de parameter aantal moet een natuurlijk getal doorgegeven worden, en aan de andere parameters moet een DNA-sequentie doorgegeven worden. De functie moet de DNA-sequentie teruggeven die is opgebouwd uit de gegeven prefix, gevolgd door een infix en de gegeven suffix, waarbij de infix bestaat uit het opgegeven aantal herhalingen van de gegeven kopie.
- Schrijf een functie kopie_aantal waaraan een DNA-sequentie $s$ moet doorgegeven worden. De functie moet een tuple bestaande uit een DNA-sequentie $k$ en een natuurlijk getal $n \in \mathbb{N}_0$ teruggeven, waarbij $k$ de kortst mogelijke DNA sequentie is waarvoor de gegeven DNA-sequentie $s$ bestaat uit $n$ herhalingen van $k$.
- Schrijf een functie LGU waaraan twee DNA-sequenties moeten doorgegeven worden. De functie heeft ook nog een optionele parameter suffix (standaardwaarde: False) waaraan een Booleaanse waarde moet doorgegeven worden. Indien de waarde False wordt doorgegeven aan de parameter suffix, dan moet de functie de langste gemeenschappelijke prefix (langste gemeenschappelijke string aan het begin van de gegeven sequenties) teruggeven. Indien de waarde True wordt doorgegeven aan de parameter suffix, dan moet de functie de langste gemeenschappelijke suffix (langste gemeenschappelijke string aan het einde van de gegeven sequenties) teruggeven.
- Gebruik nu de voorgaande twee functies om een functie CNV
te schrijven waaraan twee DNA-sequenties moeten doorgegeven worden.
Indien de twee DNA-sequenties eenzelfde prefix en suffix hebben, en
enkel van elkaar verschillen in het aantal tussenliggende herhalingen
van dezelfde kopie, dan moet de functie een tuple teruggeven dat bestaat
uit de (kortst mogelijke) kopie en het aantal herhalingen van die kopie
in de eerste en in de tweede sequentie. Anders moet de functie een AssertionError opwerpen met de
boodschap geen CNV gevonden.
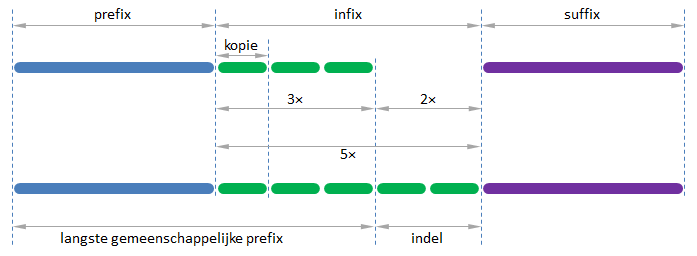
Gebruik de volgende procedure om de verschillende onderdelen te
identificeren waaruit de twee gegeven DNA-sequenties zijn opgebouwd (zie
ook onderstaande figuur):
- controleer dat de sequenties verschillend zijn, anders geen CNV gevonden
- bepaal de langste gemeenschappelijk prefix (LGP) van de sequenties
- bepaal de suffix als het resterende deel van de kortste sequentie (het deel dat niet behoort tot de LGP)
- controleer dat de langste sequentie eindigt met de gevonden suffix, anders geen CNV gevonden
- bepaal de indel als het deel tussen de LGP en de suffix in de langste sequentie
- bepaal de kortst mogelijke kopie en het aantal herhalingen van die kopie waaruit de indel is opgebouwd (bij CNV bestaat de indel immers uit één of meer herhalingen van de kopie)
- bepaal het aantal extra herhalingen van de kopie op het einde van de LGP
Voorbeeld
>>> repliceer('GATC') 'GATC' >>> repliceer('GATC', aantal=4) 'GATCGATCGATCGATC' >>> repliceer('GATC', aantal=2, prefix='TAGCC') 'TAGCCGATCGATC' >>> repliceer(kopie='GATC', aantal=3, suffix='AAGCTC') 'GATCGATCGATCAAGCTC' >>> repliceer(kopie='GATC', aantal=3, prefix='TAGCC', suffix='AAGCTC') 'TAGCCGATCGATCGATCAAGCTC' >>> repliceer(kopie='GATC', aantal=5, suffix='AAGCTC', prefix='TAGCC') 'TAGCCGATCGATCGATCGATCGATCAAGCTC' >>> kopie_aantal('CTCTCTCTCTCTCTCTCTCTCTCT') # repliceer(kopie='CT', aantal=12) ('CT', 12) >>> kopie_aantal('GATCGATCGATCGATC') # repliceer(kopie='GATC', aantal=4) ('GATC', 4) >>> kopie_aantal(repliceer('GATCGATC', aantal=2)) ('GATC', 4) >>> kopie_aantal('GATCGATCGATCGATCG') # repliceer(kopie='GATC', aantal=4) + 'G' ('GATCGATCGATCGATCG', 1) >>> seq1 = 'TAGCCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=3, prefix='TAGCC', suffix='AAGCTC') >>> seq2 = 'TAGCCGATCGATCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=5, suffix='AAGCTC', prefix='TAGCC') >>> LGU(seq1, seq2) 'TAGCCGATCGATCGATC' >>> LGU(seq1, seq2, suffix=True) 'CGATCGATCGATCAAGCTC' >>> LGU(seq1, seq1) 'TAGCCGATCGATCGATCAAGCTC' >>> seq1 = 'TAGCCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=3, prefix='TAGCC', suffix='AAGCTC') >>> seq2 = 'TAGCCGATCGATCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=5, suffix='AAGCTC', prefix='TAGCC') >>> CNV(seq1, seq2) ('GATC', 3, 5) >>> CNV(seq2, seq1) ('GATC', 5, 3) >>> CNV(seq1, seq1) Traceback (most recent call last): AssertionError: geen CNV gevonden >>> seq3 = 'TAGCGATCGATCGATCGATCGATCAAGCTC' # repliceer(kopie='GATC', aantal=5, prefix='TAGC', suffix='AAGCTC') >>> CNV(seq1, seq3) ('GATCGAT', 0, 1) >>> seq4 = 'TAGCCGATCGATCGATCGATCGATCAGCTC' # repliceer(kopie='GATC', aantal=5, suffix='AGCTC', prefix='TAGCC') >>> CNV(seq1, seq4) Traceback (most recent call last): AssertionError: geen CNV gevonden
| Added by: | Peter Dawyndt |
| Date: | 2015-01-02 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |



 RSS
RSS