| Submit | All submissions | Best solutions | Back to list |
PROG0525 - Protein codes |
For centuries, people have been looking for secret messages that are hidden in books, songs played backward, funny-looking Martian mensas, or some other objects. Some believe that hidden messages in the Bible cannot be just coincidence — they must have been put in there deliberately by God himself.
The hunt for hidden messages has been popularized in modern times by the book The Bible Code. It was written by American journalist Michael Drosnin, who claims that the Hebrew Bible contains a very complex code that reveals events that took place thousands of years after the Bible was written. Drosnin contends that some foretold events later happened exactly as predicted. Drosnin asserts that the Bible also contains hidden messages that predict the future.
To find hidden messages, Drosnin makes use of a simple technique: start at a given letter in a text and repeatedly step a fixed number of letters forward of backward. Consider the verse in the Book of Genesis (King James Version): 31:28 And hast not suffered me to kiss my sons and my daughters? Thou hast now done foolishly in so doing. If you start at the R in daughters, and skip over three letters to the O in thou, and three more to the S in hast, and so on, the hidden message Roswell is revealed! A companion hidden message — UFO — is found by starting at the U in thou, and repeatedly stepping forward 12 letters. This can't be a coincidence, no?
The claim that no human could have encoded the Bible in this way and that the messages cannot be just coincidence, has been questioned many times. Some critics of Drosnin say the journalist is just data mining: in any text a large amount of hidden messages can be found using Drosnin's technique and a little bit of creativity. Michael Drosnin responded to the criticism in Newsweek, by stating that "When my critics find a message about the assassination of a prime minister encrypted in Moby Dick, I'll believe them". Mathematician Brendan McKay of Australian National University and his colleagues took up the challenge, and proved that this really is not all that challenging.
Assignment
Proteins are large biological molecules consisting of a long chain of amino acid residues. There are 20 amino acids that are used by living cells to build proteins, which are all represented by a capital letter (only the capitals B, J, O, U, X and Z do not correspond to an amino acid). Because the proteins that are encoded in the human genome can be represented in this was as strings of uppercase letters, Drosnin's technique is also applicable to search for hidden messages encoded in man itself.
The Latin phrase alea iacta est (the die is cast) is mainly known because it was used in 49 BC by Julias Caesar as he led his army across the river Rubicon. With this step, he entered Italy at the head of his army in defiance of the Senate and began his long civil war against Pompey and the Optimates. All words of this phrase are for example hidden in the protein sequence
HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM
which is part of a protein complex that allows us to smell. If we start at position 21 and successively skip 4 positions forward, we read the word ALEA. The same word can also be found by starting at the same position and successively skipping 11 positions forward. We may also start at position 36, and successively skip 11 positions backward to find a third occurrence of the word ALEA. The following table shows that the same protein also has two occurrences of the word IACTA and two occurrences of the word EST.
| start | step | length | word |
| 0 | 1 | 57 | HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM |
| 21 | 4 | 4 | A L E A |
| 21 | 11 | 4 | A L E A |
| 36 | -11 | 4 | A E L A |
| 27 | -6 | 5 | A T C A I |
| 27 | 6 | 5 | I A C T A |
| 29 | -10 | 3 | T S E |
| 29 | 8 | 3 | E S T |
To find similar secret messages in a given protein sequence, you may proceed as follows:
- Write a function aminoword that takes a string. The function must return a Boolean value that indicates whether or not the given string is an aminoword. A string is considered to be an aminoword is it has at least length two and only contains letters that are used as shorthand notation for an amino acid (all letters except B, J, O, U, X and Z). In determining whether a string is an amino word, the function should not make a distinction between uppercase and lowercase letters.
- Write a function positions that takes two string arguments: a protein sequence and a letter. The function must return a list containing all positions in the given protein sequence where the given letter is found. In searching for letters in the protein sequence, the function should make a distinction between uppercase and lowercase letters.
- Write a function proteincode that takes four arguments. The first argument must be a protein sequence. The next three arguments should be integers: $p \in \mathbb{N}$, $s \in \mathbb{Z}$ and $l \in \mathbb{N}$. The function must return the word of length $l$ that is read from the protein sequence by starting at position $p$ and successively skipping $s$ positions forward (or backward in case $s$ is negative). If it is not possible to read a word of length $s$ from the protein following these instructions — because the starting position does not fall within the limits of the protein or because skipping letters crosses the limits of the protein — the function must return the empty string.
- Use the previous three functions to write a function proteinsearch that takes two string arguments: a protein sequence and a string. In case the given string is not an amino word (according to the definition of the function aminoword), the function must raise an AssertionError with the message invalid aminoword. Otherwise, the function must return a list containing all possible ways the given amino word can be read from the given protein sequence. A way of reading is represented as the tuple ($p$, $s$), where $p$ indicates the starting position and $s$ indicates the number of positions that must be skipped forward or backward. The tuples must be sorted in increasing order in the returned list. Implement the following procedure to find all ways a given amino word can be read from a given protein sequence:
- find all positions in the protein where the first letter of the amino word is found
- find all positions in the protein where the second letter of the amino word is found
- each combination of a possible position of the first and the second letter, also defines the number of positions that must be skipped forward or backward in order to reach the second letter when starting from the first letter
- check for each combination if you can read the given amino word when starting from the position of the first letter and successively skipping the computed number of positions forward or backward
- ga voor elke combinatie na of je door vanaf de startpostie (positie van de eerste letter) herhaaldelijk het gevonden aantal posities vooruit of achteruit te springen, het gegeven aminowoord kunt uitlezen
Example
>>> aminoword('ALEA') True >>> aminoword('iacta') True >>> aminoword('Proline') False >>> protein = 'HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM' >>> positions(protein, 'A') [3, 21, 28, 33, 36, 51, 54] >>> positions(protein, 't') [8, 9, 18, 41, 45] >>> proteincode(protein, 21, 11, 4) 'ALEA' >>> proteincode(protein, 27, -6, 5) 'IACTA' >>> proteincode(protein, 29, 8, 3) 'EST' >>> proteincode(protein, 0, 25, 6) '' >>> protein = 'HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM' >>> proteinsearch(protein, 'ALEA') [(21, 4), (21, 11), (36, -11)] >>> proteinsearch(protein, 'iacta') [(27, -6), (27, 6)] >>> proteinsearch(protein, 'EST') [(29, -10), (29, 8)] >>> proteinsearch(protein, 'EST', maxstep=8) [(29, 8)] >>> proteinsearch(protein, 'Proline') Traceback (most recent call last): AssertionError: invalid amino word Traceback (most recent call last): AssertionError: invalid amino word
De mens is al eeuwenlang op zoek naar geheime boodschappen die verborgen zitten in boeken, achterwaarts afgespeelde muziek, vreemd uitziende plateau's op Mars, of andere dingen. Sommigen geloven bijvoorbeeld stellig dat er verborgen boodschappen in de Bijbel zitten die niet op toeval kunnen berusten — ze moeten er wel moedwillig in aangebracht zijn door niemand minder dan God de Vader zelf.
De zoektocht naar verborgen boodschappen werd nieuw leven ingeblazen door de publicatie van het boek The Bible Code. Het is geschreven door journalist Michael Drosnin, die beweert dat de Hebreeuwse Bijbel een zeer complexe code bevat die gebeurtenissen onthult die plaatsvonden duizenden jaren nadat de Bijbel werd geschreven. Volgens Drosnin is het dan ook logisch dat er in de Bijbel boodschappen verborgen moeten zitten die iets te vertellen hebben over onze toekomst.
Om verborgen boodschappen te vinden, maakt Drosnin gebruik van een eenvoudige techniek: start bij een bepaalde letter in een tekst, en spring daarna telkens een vast aantal letters vooruit of achteruit. Op die manier valt bijvoorbeeld in bijbelvers Genesis 31:28 (King James versie) "And hast not suffered me to kiss my sons and my daughters? Thou hast now done foolishly in so doing." het woord Roswell te lezen door te starten vanaf de R in daughters, drie letters vooruit te springen naar de O in thou, nog drie letters vooruit te springen naar de S in hast, enzoverder. In datzelfde vers lees je ook het woord UFO door te starten vanaf de U in thou, en daarna telkens 12 letters vooruit te springen. En dat kan toch zeker geen toeval zijn?
De bewering dat de verborgen boodschappen in de Bijbel onmogelijk aan toeval toe te schrijven zijn, werd echter vanuit allerlei hoeken in vraag gesteld. Sommige critici van Drosnin zeggen dat de journalist niets anders doet dan data mining: in elke willekeurige tekst vallen met de gebruikte techniek en een gezonde dosis creativiteit een groot aantal verborgen boodschappen te vinden. Michael Drosnin reageerde in Newsweek op de kritiek door te stellen dat "When my critics find a message about the assassination of a prime minister encrypted in Moby Dick, I'll believe them". Wiskundige Brendan McKay van de Australian University nam de handschoen op, en toonde aan dat dat helemaal geen probleem bleek te zijn.
Opgave
Eiwitten bestaan uit polymere ketens van aminozuren. De eiwitten die aangemaakt worden in cellen van levende organismen zijn opgebouwd uit 20 verschillende aminozuren, die elk voorgesteld worden door een hoofdletter (enkel de hoofdletters B, J, O, U, X en Z corresponderen niet met een aminozuur). Omdat de eiwitten die gecodeerd zitten in het menselijk genoom op die manier kunnen voorgesteld worden als strings van hoofdletters, kunnen we de methode van Drosnin ook toepassen om op zoek te gaan naar geheime boodschappen die gecodeerd zitten in de mens zelf.
De Latijnse spreuk alea iacta est (de teerling is geworpen) kennen we vooral omdat ze in 49 voor Christus door Julias Caesar werd uitgesproken, toen die met een staand leger de Rubicon overstak om een staatsgreep te plegen in Rome. De woorden van deze zin zitten bijvoorbeeld verborgen in de eiwitsequentie
HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM
die deel uitmaakt van een complexe reeks eiwitten die er samen voor zorgen dat we kunnen ruiken. Als we starten op positie 21, en telkens 4 posities vooruit springen, dan lezen we het woord ALEA. Dit zelfde woord lees je door op dezelfde positie te starten, en telkens 11 posities vooruit te springen. Je kunt ook starten op positie 36, en telkens 11 posities achteruit springen om het woord ALEA nog een derde keer terug te vinden in het eiwit. Onderstaande tabel geeft aan dat er in het eiwit ook nog twee voorkomens van het woord IACTA en twee voorkomens van het woord EST te vinden zijn.
| start | stap | lengte | woord |
| 0 | 1 | 57 | HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM |
| 21 | 4 | 4 | A L E A |
| 21 | 11 | 4 | A L E A |
| 36 | -11 | 4 | A E L A |
| 27 | -6 | 5 | A T C A I |
| 27 | 6 | 5 | I A C T A |
| 29 | -10 | 3 | T S E |
| 29 | 8 | 3 | E S T |
Om dergelijke geheime boodschappen te vinden in een gegeven eiwitsequentie ga je als volgt te werk:
- Schrijf een functie aminowoord waaraan een string moet doorgegeven worden. De functie moet een Booleaanse waarde teruggeven, die aangeeft of de gegeven string een aminowoord is of niet. Een string is een aminowoord als het minstens lengte twee heeft en enkel bestaat uit letters die als afkorting voor aminozuren gebruikt worden (alle letters behalve B, J, O, U, X en Z). Om te bepalen of een string een aminowoord is, mag de functie geen onderscheid maken tussen hoofdletters en kleine letters.
- Schrijf een functie posities waaraan twee stringargumenten moeten doorgegeven worden: een eiwitsequentie en een letter. De functie moet een lijst teruggeven van alle posities in de gegeven eiwitsequentie waar de gegeven letter terug te vinden is. Bij het zoeken naar letters in een eiwitsequentie mag de functie geen onderscheid maken tussen hoofdletters en kleine letters.
- Schrijf een functie eiwitcode waaraan vier argumenten moeten doorgegeven worden. Het eerste argument moet een eiwitsequentie zijn. De volgende drie argumenten moeten getallen zijn: $p \in \mathbb{N}$, $s \in \mathbb{Z}$ en $l \in \mathbb{N}$. De functie moet het woord van lengte $l$ teruggeven dat kan uitgelezen worden uit de gegeven eiwitsequentie, door te starten op positie $p$ en telkens $s$ posities vooruit (of achteruit indien $s$ negatief is) te springen. Indien het niet mogelijk is om op de aangegeven manier een woord van lengte $l$ uit het eiwit te lezen — omdat de startpositie niet binnen de grenzen van het eiwit ligt, of omdat je bij het springen voorbij de grenzen van het eiwit springt — dan moet de functie de lege string teruggeven.
- Gebruik de drie vorige functies om een functie eiwitzoeker te schrijven waaraan een eiwitsequentie en een string moeten doorgegeven worden. Indien de gegeven string geen aminowoord is (volgens de definitie van de functie aminowoord), dan moet de functie een AssertionError opwerpen met de boodschap ongeldig aminowoord. Anders moet de functie een lijst teruggeven met alle manieren waarop het aminowoord uit de eiwitsequentie kan gelezen worden. Een manier van uitlezen wordt voorgesteld als een tuple ($p$, $s$), waarbij $p$ de startpositie aangeeft vanaf waar men start met lezen, en $s$ hoeveel posities er telkens vooruit of achteruit moet gesprongen worden. De tuples moeten in oplopende volgorde voorkomen in de lijst die wordt teruggegeven. Implementeer de volgende procedure om alle manieren te vinden waarop een gegeven aminowoord uit een gegeven eiwitsequentie kan gelezen worden:
- zoek alle posities in het eiwit waarop de eerste letter van het aminowoord voorkomt
- zoek alle posities in het eiwit waarop de tweede letter van het aminowoord voorkomt
- elke combinatie van een mogelijke positie van de eerste en de tweede letter legt vast hoeveel posities je vooruit of achteruit moet springen om de tweede letter te bereiken vanaf de eerste letter
- ga voor elke combinatie na of je het gegeven aminowoord kunt uitlezen te starten vanaf de positie van de eerste letter en herhaaldelijk het berekende aantal posities vooruit of achteruit te springen
Voorbeeld
>>> aminowoord('ALEA') True >>> aminowoord('iacta') True >>> aminowoord('Proline') False >>> eiwit = 'HGLAVPFRTTHPSLECGRTSWARWSLDIAEFWLAWEASDCITDEDTKFQGDAVVAQM' >>> posities(eiwit, 'A') [3, 21, 28, 33, 36, 51, 54] >>> posities(eiwit, 'l') [2, 13, 25, 32] >>> eiwitcode(eiwit, 21, 11, 4) 'ALEA' >>> eiwitcode(eiwit, 27, -6, 5) 'IACTA' >>> eiwitcode(eiwit, 29, 8, 3) 'EST' >>> eiwitcode(eiwit, 0, 25, 6) '' >>> eiwitzoeker(eiwit, 'ALEA') [(21, 4), (21, 11), (36, -11)] >>> eiwitzoeker(eiwit, 'iacta') [(27, -6), (27, 6)] >>> eiwitzoeker(eiwit, 'EST') [(29, -10), (29, 8)] >>> eiwitzoeker(eiwit, 'EST', maxstap=8) [(29, 8)] >>> eiwitzoeker(eiwit, 'Proline') Traceback (most recent call last): AssertionError: ongeldig aminowoord
| Added by: | Peter Dawyndt |
| Date: | 2015-01-06 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |




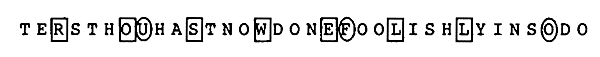
 RSS
RSS