| Submit | All submissions | Best solutions | Back to list |
PROG0366 - Chromosomal crossover |
As one of the last steps of genetic recombination two homologous chromosomes can exchange genetic material during meiosis in a process that is referred to as synapsis. Because of this chromosomal crossover, recombined chromosomes arise. Crossover usually occurs when corresponding regions of corresponding chromosomes break and thereafter attach back to the other chromosome.
A theoretical description of crossover was introduced by Thomas Hunt Morgan, who was inspired by the work of the Belgian Professor Frans Alfons Janssens of the University of Leuven. The latter had already described his chiasmatype theory in 1909.
Assignment
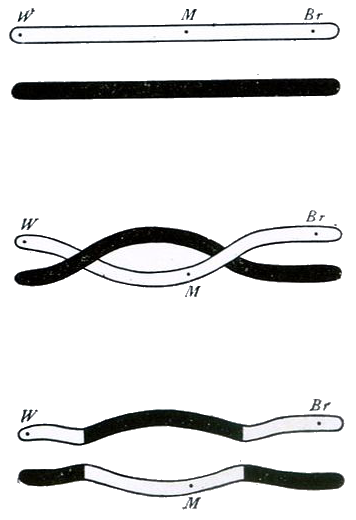
In this assignment, we represent a chromosome as a list or a tuple of strictly increasing integers (each number is larger than the previous number). The points where two chromosomes can break open - and where a crossover can occur - are indicated by the common numbers in the two corresponding lists or tuples. This is illustrated in the figure below, where crossover can occur where the two chromosomes meet.
The chromosomes can be navigated through the following way:
- As a starting point the beginning (the left side) of one of the two chromosomes can be selected.
- Stepping forward (to the right) is possible from any point.
- At a crossover point, you have the choice to continue on the same chromosome, or switch to the other chromosome.
Asked:
- Write a function crossoverpoints to which two chromosomes must be passed. The function should return how many crossover points there are between these chromosomes.
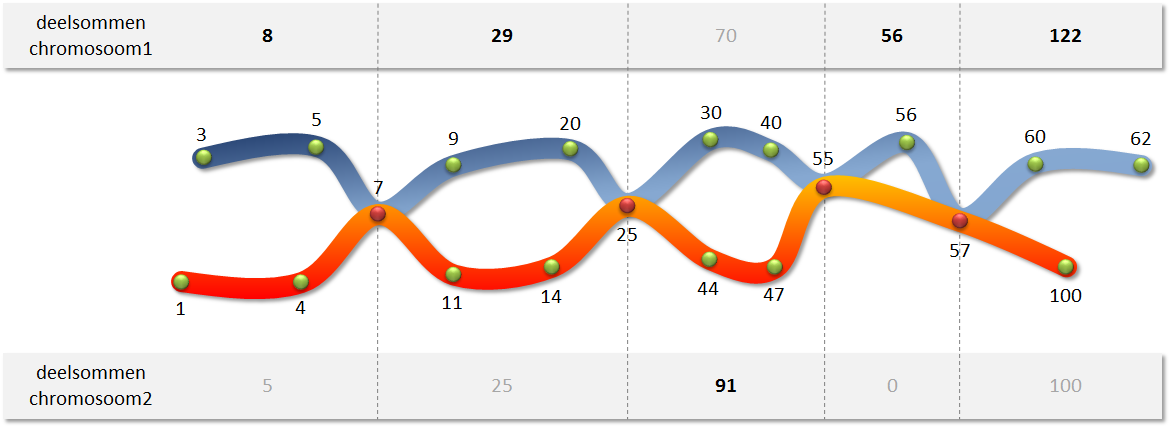
- Write a function maximumSum to which two chromosomes must be passed. The function should determine the maximum sum of all paths that can be followed in the manner described above. In the example above, the maximum sum of 450 is achieved by going through the following path:
3 + 5 + 7 + 9 + 20 + 25 + 44 + 47 + 55 + 56 + 57 + 60 + 62The maximum sum is found by determining the maximum partial sums of the fragments before the first crossover point, between successive crossover points, and after the last crossover point. Note that it is possible that two partial sums are equal, if the end points themselves are crossover points, or if two crossover points immediately follow each other without intermediate points.
Algorithm
In order to find the crossover points, you can use the fact that the numbers in the two data frames are strictly increasing. In that way, you can go through the lists simultaneously from left to right, while you keep track of your position for each list separately. Always move one step ahead in the list from where at the current position is the smallest number. If you find a similar number on the current position in both lists, then you have found a crossover point. While running through both lists, you can also determine the partial sums before, between, and after the crossover points.
Example
>>> chromosome1 = [3, 5, 7, 9, 20, 25, 30, 40, 55, 56, 57, 60, 62] >>> chromosome2 = [1, 4, 7, 11, 14, 25, 44, 47, 55, 57, 100] >>> crossoverpoints(chromosome1, chromosome2) 4 >>> maximumSum(chromosome1, chromosome2) 450 >>> chromosome1 = [-5, 100, 1000, 1005] >>> chromosoom2 = [-12, 1000, 1001] >>> crossoverpoints(chromosome1, chromosome2) 1 >>> maximumSum(chromosome1, chromosome2) 2100
Source
Morgan TH (1916). A
Critique of the Theory of Evolution. Princeton, NJ, Princeton University
Press. ![]()
Als één van de laatste stappen van genetische recombinatie kunnen twee homologe chromosomen tijdens de meiose genetisch materiaal uitwisselen in een proces dat synapsis genoemd wordt. Door deze chromosomale crossover ontstaan gerecombineerde chromosomen. Crossover komt meestal voor wanneer overeenkomstige regio's van overeenkomstige chromosomen breken en zich daarna terug aanhechten aan het andere chromosoom.
Een theoretische beschrijving van crossover werd geïntroduceerd door Thomas Hunt Morgan, die zich daarbij liet inspireren op het werk van de Belgische professor Frans Alfons Janssens van de Katholieke Universiteit Leuven. Deze laatste had het fenomeen reeds in 1909 beschreven en noemde het chiasmatypie.
Opgave
In deze opgave stellen we een chromosoom voor als een lijst of een tuple van strikt stijgende gehele getallen (elk getal is dus groter dan het voorgaande getal). De punten waar twee chromosomen kunnen openbreken — en waar zich dus crossover kan voordoen — worden aangegeven door de gemeenschappelijke getallen in de twee corresponderende lijsten of tuples. Dit wordt geïllustreerd in onderstaande afbeelding, waarbij crossover kan voorkomen waar de twee chromosomen elkaar raken.
De chromosomen kunnen op de volgende manier doorlopen worden:
- Als startpunt kan het begin (de linkerkant) van één van beide chromosomen gekozen worden.
- Vanaf elk punt kan vooruit (naar rechts) gestapt worden.
- Bij een crossoverpunt heb je de keuze om verder te stappen op hetzelfde chromosoom, of over te schakelen op het andere chromosoom.
Gevraagd wordt:
- Schrijf een functie crossoverpunten waaraan twee chromosomen moeten doorgegeven worden. De functie moet teruggeven hoeveel crossoverpunten er zijn tussen deze chromosomen.
- Schrijf een functie maximaleSom waaraan twee chromosomen moeten doorgegeven worden. De functie moet de maximale som bepalen van alle paden die op de hierboven beschreven manier kunnen doorlopen worden. In het bovenstaand voorbeeld wordt de maximale som 450 bereikt door het volgende pad te doorlopen:
3 + 5 + 7 + 9 + 20 + 25 + 44 + 47 + 55 + 56 + 57 + 60 + 62De maximale som wordt gevonden door de maximale deelsommen te bepalen van de fragmenten vóór het eerste crossoverpunt, tussen opeenvolgende crossoverpunten, en na het laatste crossoverpunt. Merk op dat het mogelijk is dat twee deelsommen gelijk zijn indien de eindpunten zelf crossoverpunten zijn, of indien twee crossoverpunten elkaar onmiddellijk opvolgen zonder tussenliggende punten.
Algoritme
Om de crossoverpunten te vinden, kan je gebruik maken van het feit dat de getallen in de twee gegeven lijsten strikt stijgend zijn. Je kunt beide lijsten dan tegelijkertijd van links naar rechts doorlopen, waarbij je voor elke lijst afzonderlijk bijhoudt op welke positie je je momenteel bevindt. Ga telkens één stap vooruit in de lijst waar op de huidige positie het kleinste getal staat. Als je op de huidige positie in beide lijsten eenzelfde getal aantreft, dan heb je een crossoverpunt gevonden. Tijdens het doorlopen van beide lijsten kan je meteen ook al de deelsommen voor, tussen, en na de crossoverpunten bepalen.
Voorbeeld
>>> chromosoom1 = [3, 5, 7, 9, 20, 25, 30, 40, 55, 56, 57, 60, 62] >>> chromosoom2 = [1, 4, 7, 11, 14, 25, 44, 47, 55, 57, 100] >>> crossoverpunten(chromosoom1, chromosoom2) 4 >>> maximaleSom(chromosoom1, chromosoom2) 450 >>> chromosoom1 = [-5, 100, 1000, 1005] >>> chromosoom2 = [-12, 1000, 1001] >>> crossoverpunten(chromosoom1, chromosoom2) 1 >>> maximaleSom(chromosoom1, chromosoom2) 2100
Bronnen
Morgan TH (1916). A
Critique of the Theory of Evolution. Princeton, NJ, Princeton University
Press. ![]()
| Added by: | Peter Dawyndt |
| Date: | 2013-04-03 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS