| Submit | All submissions | Best solutions | Back to list |
PROG0287 - Chaos game |
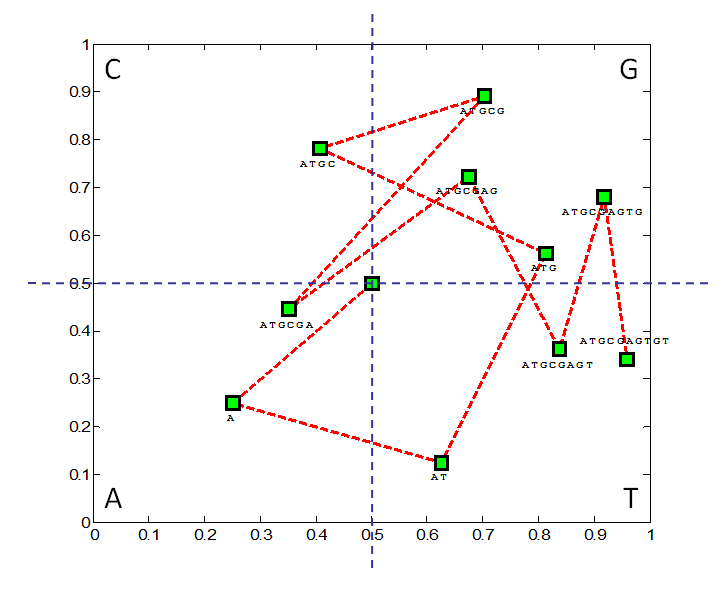
Chaos Game Representation (CGR) is a graphical way of representing a DNA sequence, in such a way that certain patterns in the nucleotide sequence become immediate clear at a glance. The representation is done within the unit square, in which the four corner points are labeled with the four nucleotides: the point (0, 0) corresponds to the letter a, the point (0, 1) to the letter c, the point (1, 1) to the letter g and the point (1, 0) to the letter t. The starting point for each CGR is always the point (0.5, 0.5). Thereafter, for each letter of the nucleotide sequence the next point is computed as the point that is half-way on the line that connects the last point to the corner point that is labeled with the nucleotide. The figure below shows an example of the CGR that is built from the DNA sequence atgcgagtgt.
Input
A number of DNA sequences (strings that only contain the letters a, c, g and t) that are each on their own line. The list of DNA sequences is terminated with a line containing the word stop.
Output
For each DNA sequence from the input, the $x$ and the $y$ co-ordinate of the last point of the corresponding CGR should be printed on a separate line.
Example
Input:
atgcgagtgt tcttacaggtaagggggatta tctcagcgggagaagctcagttaggacgcgaaacccg gactgcgccaatcgaacatacaaagtttggtct tagtaaggtgaaagggaggccagaggtcggctcct stop
Output:
0.95947265625 0.34033203125 0.435980558395 0.0607464313507 0.505093238589 0.945012472414 0.74807859509 0.345834313601 0.589490680417 0.435239574072
Chaos Game Representation (CGR) is een grafische manier om een DNA sequentie voor te stellen, zodat bepaalde patronen in de nucleotidesequentie in één oogopslag kunnen herkend worden. De voorstelling gebeurt binnen het eenheidsvierkant, waarbij de vier hoekpunten gelabeld worden met de vier nucleotiden: het punt (0, 0) komt overeen met de letter a, het punt (0, 1) met de letter c, het punt (1, 1) met de letter g en het punt (1, 0) met de letter t. Het vertrekpunt van elke CGR is steeds het punt (0.5, 0.5). Daarna wordt voor elke letter van de nucleotidesequentie een volgende punt berekend dat halverweg ligt op de lijn die het laatste punt verbindt met het hoekpunt dat correspondeert met die letter. Onderstaande figuur toont bijvoorbeeld hoe de CGR voor de DNA sequentie atgcgagtgt wordt opgebouwd.
Invoer
Een aantal DNA sequenties (strings die enkel bestaan uit de letters a, c, g en t) die elk op een afzonderlijke regel staan. De lijst van DNA sequenties wordt afgesloten met een regel met daarop de tekst stop.
Uitvoer
Schrijf voor elke DNA sequentie uit de invoer de $x$- en de $y$-coördinaat van het laatste punt van de corresponderende CGR uit, elk op een afzonderlijke regel.
Voorbeeld
Invoer:
atgcgagtgt tcttacaggtaagggggatta tctcagcgggagaagctcagttaggacgcgaaacccg gactgcgccaatcgaacatacaaagtttggtct tagtaaggtgaaagggaggccagaggtcggctcct stop
Uitvoer:
0.95947265625 0.34033203125 0.435980558395 0.0607464313507 0.505093238589 0.945012472414 0.74807859509 0.345834313601 0.589490680417 0.435239574072
| Added by: | Peter Dawyndt |
| Date: | 2012-09-16 |
| Time limit: | 10s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS