| Submit | All submissions | Best solutions | Back to list |
PROG0259 - Protein folding |
A notorious problem in molecular biology,is protein folding. A protein molecule consists of a chain of amino acids that spontaneously folds into a three-dimensional shape that is more energetically favorable . This binding energy depends on the mutual position of all amino acids. This provides so many options that even the most advanced supercomputers still crash here.
In order to make the problem somewhat easier we are going to simulate a protein molecule that folds in two dimensions. The initial state is a square grid in which each amino acid has a positive or negative charge. The folding is done by placing baffles between the boxes in such a way that a circuit is created through the grid that includes all the booths. The ends of the molecule do not have to be situated at the edge of the grid.
Whenever a baffle is positioned between two boxes, the molecule is folded and the amino acids are touching each other. If there is a baffle between a box with charge $-3$ and a box with charge $4$, this results in a binding energy of $-3 \times 4 = -12$. An amino acid can affect several other amino acids, and then yield binding energy with every neighbour. Of course, due to the minus signs, binding energy can be both positive and negative.
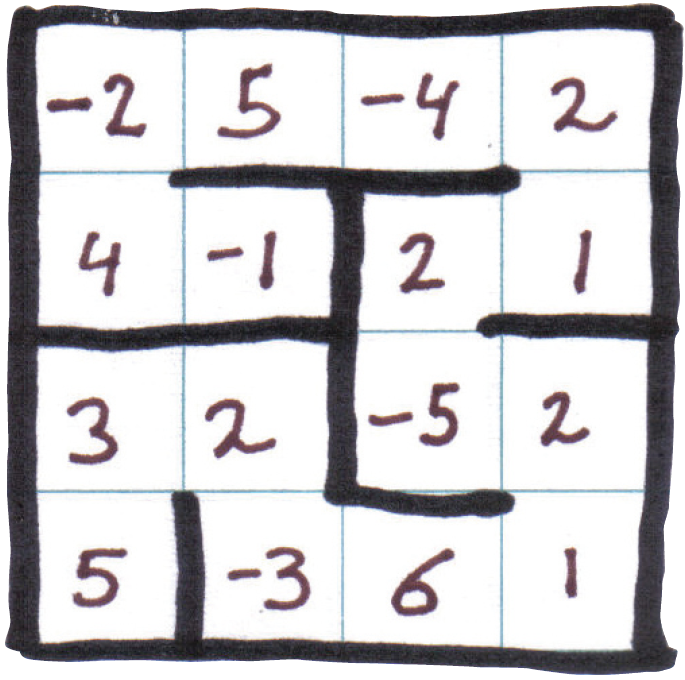
For example, you can see a $4\times4$ grid above, in which a protein with
16 amino acids is folded to a configuration with binding energy equal to
\[ \begin{multline}
\phantom{xxxxxxxxxxxx}(-1 \times 2) + (2 \times -5) + (5 \times -3) + (4
\times 3) + (5\times -1) + \phantom{x}\\
(-1 \times 2) + (-4 \times 2) + (-5 \times 6) + (1 \times 2) =
-58\phantom{xxxxxxxxxxxx} \nonumber
\end{multline} \]
Assignment
Define a class Protein with the following methods:
- An initializing method __init__ which can be used to make a protein with a given initial state. This initial stated must be given to the initializing method as a first, obligatory parameter, in the format of a list of whole numbers. The initializing method must verify whether a square grid can be made from this list. If this is not the case, an AssertionError must occur as indicated in the example below. Otherwise, the initial state must be saved in a the newly made Protein object.
- A method fold with an optional parameter with which a fold configuration for the protein can be given in the format of a list of tuples. In that list, every tuple has the format ($r_1, k_1, r_2, k_2$) and indicates that a baffle must be placed between two neighbouring box $(r_1, k_1)$ and $(r_2, k_2)$ when folding. Here, $r$ and $k$ respectively represent a row number and a column number, where the numbering always starts from aero. The method may assume that a list of tuples is given as fold configuration, and that every tuple consists of four whole numbers. The method does have to verify for every tuple if these four numbers represent two valid positions of boxes in the square grid, and that the first box is situated either on the left of the second box in the grid, or directly above. Look at the example below to see how the method must react if an invalid fold configuration was given. If a valid value was given to this optional parameter, this configuration must be kept by the Protein object. A possible former configuration is then overwritten by the new configuration. If no value was given to the optional parameter, the protein is not folded (in other words, there are no boxes).
- In order to immediately built a Protein object with a given initial state and an initial folding configuration, a folding configuration can be given to the initializing method __init__ as a second optional parameter. This has the same format and meaning of the parameter from the method fold. If a value is given for this parameter, the folding configuration must be set for the protein by calling the method fold. Otherwise, no folding configuration is initially set for the protein.
- A method bindingenergy that prints the binding energy of the protein. This binding energy must be calculated based on the folding configuration that was set for the protein. If no folding configuration was set, 0 (zero) must be printed.
- A method __str__ which can be used to print the string representation of the protein of the format of the example below. Here, the charges of the amino acids must be shown centrated over four positions in the boxes. If a folding configuration was set, the baffles must be shown in the right place.
Example
>>> protein = Protein([-2, 5, -4, 2, 4, -1, 2]) Traceback (most recent call last): AssertionError: invalid initial state >>> protein = Protein([-2, 5, -4, 2, 4, -1, 2, 1, 3, 2, -5, 2, 5, -3, 6, 1]) >>> eiwit.bindingenergy() 0 >>> print(protein) +----+----+----+----+ | -2 5 -4 2 | + + + + + | 4 -1 2 1 | + + + + + | 3 2 -5 2 | + + + + + | 5 -3 6 1 | +----+----+----+----+ >>> protein.fold([(1, 1, 1, 2), (2, 1, 2, 2), (3, 0, 3, 1), ... (1, 0, 2, 0), (0, 1, 1, 1), (1, 1, 2, 1), ... (0, 2, 1, 2), (2, 2, 3, 2), (1, 3, 2, 3)]) >>> protein.bindingenergy() -58 >>> print(protein) +----+----+----+----+ | -2 5 -4 2 | + +----+----+ + | 4 -1 | 2 1 | +----+----+ +----+ | 3 2 | -5 2 | + + +----+ + | 5 | -3 6 1 | +----+----+----+----+ >>> protein.fold([(1, 1, 1, 2), (2, 1, 2, 2), (4, 0, 4, 1)]) Traceback (most recent call last): AssertionError: invalid configuration >>> protein.fold([(1, 1, 1, 2), (2, 1, 3, 3), (3, 0, 3, 1)]) Traceback (most recent call last): AssertionError: invalid configuration >>> protein = Protein([7, -2, -4, 3], [(0, 1, 1, 1)]) >>> protein.bindingenergy() -6 >>> print(protein) +----+----+ | 7 -2 | + +----+ | -4 3 | +----+----+
Een berucht probleem in de moleculaire biologie is het vouwen van eiwitten. Een eiwitmolecuul bestaat uit een keten van aminozuren die zichzelf spontaan opvouwt tot een driedimensionale vorm die energetisch het gunstigst is. Deze bindingsenergie hangt af van de onderlinge positie van alle aminozuren. Dit levert zoveel mogelijkheden op dat zelfs de modernste supercomputers hier nog op stuklopen.
Om het probleem iets makkelijker te maken, gaan we een eiwitmolecuul simuleren dat zich in twee dimensies opvouwt. De begintoestand is een vierkant rooster waarin elk aminozuur een positieve of negatieve lading heeft. Het opvouwen gebeurt door schotjes tussen de hokjes te plaatsen, zodanig dat een keten door het rooster ontstaat die alle hokjes omvat. De uiteinden van het molecuul hoeven niet aan de rand van het rooster te liggen.
Overal waar een schotje tussen twee hokjes staat, is het molecuul gevouwen en raken de aminozuren elkaar. Als tussen een hokje met lading $-3$ en een hokje met lading $4$ een schotje staat, dan levert dit een bindingsenergie van $-3 \times 4 = -12$ op. Een aminozuur kan aan meerdere aminozuren raken, en levert dan met elke buur bindingsenergie op. Uiteraard kan, vanwege de mintekens, bindingsenergie zowel positief als negatief zijn.
Als voorbeeld staat hierboven een $4\times4$ rooster, waarin een eiwit
met 16 aminozuren is opgevouwen tot een configuratie met bindingsenergie
gelijk aan \[ \begin{multline}
\phantom{xxxxxxxxxxxx}(-1 \times 2) + (2 \times -5) + (5 \times -3) + (4
\times 3) + (5\times -1) + \phantom{x}\\
(-1 \times 2) + (-4 \times 2) + (-5 \times 6) + (1 \times 2) =
-58\phantom{xxxxxxxxxxxx} \nonumber
\end{multline} \]
Opgave
Definieer een klasse Eiwit met volgende methoden:
- Een initialisatiemethode __init__ waarmee een eiwit kan aangemaakt worden met gegeven begintoestand. Deze begintoestand moet als eerste verplichte parameter aan de initialisatiemethode doorgegeven worden. Dit onder de vorm van een lijst van gehele getallen. De initialisatiemethode moet nagaan of met deze lijst een vierkant rooster kan gevormd worden waarin de getallen van links naar rechts en van onder naar boven ingevuld worden. Indien dat niet het geval is, dan moet er een AssertionError opgeworpen worden zoals aangegeven in onderstaande voorbeeld. Anders moet de begintoestand bijgehouden worden in het nieuw aangemaakte Eiwit object.
- Een methode opvouwen met een optionele parameter waarmee een vouwconfiguratie voor het eiwit kan doorgeven worden onder de vorm van een lijst van tuples. Daarin is elk tuple van de vorm ($r_1, k_1, r_2, k_2$) en geeft aan dat er bij het opvouwen een schotje moet geplaatst worden tussen de twee aangrenzende hokjes $(r_1, k_1)$ en $(r_2, k_2)$. Hierbij staan $r$ en $k$ respectievelijk voor een rij- en kolomnummer, waarbij de nummering telkens start vanaf nul. De methode mag ervan uitgaan dat als vouwconfiguratie een lijst van tuples wordt doorgegeven, en dat elk van deze tuples bestaat uit vier gehele getallen. Voor elk tuple moet de methode wel nagaan dat deze vier getallen staan voor twee geldige posities van hokjes in het vierkant rooster, en dat het eerste hokje links van het tweede hokje staat in het rooster, of er vlak boven. Bekijk onderstaand voorbeeld om te zien hoe de methode moet reageren als geen geldige vouwconfiguratie werd opgegeven. Als er een geldige waarde voor de optionele parameter werd opgegeven, dan moet deze configuratie bijgehouden worden door het Eiwit object. Een vorige configuratie wordt hierbij eventueel overschreven. Als er geen waarde voor de optionele parameter opgegeven wordt, dan moet ingesteld worden dat het eiwit niet opgevouwen is (er zijn met andere woorden geen hokjes).
- Om onmiddellijk een Eiwit object te kunnen aanmaken met gegeven begintoestand en initiële vouwconfiguratie, kan aan de initialisatiemethode __init__ ook een vouwconfiguratie als tweede optionele parameter doorgegeven worden. Deze heeft dezelfde vorm en betekenis als de parameter van de methode opvouwen. Indien een waarde voor deze parameter wordt opgegeven, dan moet een vouwconfiguratie ingesteld worden voor het eiwit door de methode opvouwen aan te roepen. Anders is er initieel geen vouwconfiguratie ingesteld voor het eiwit.
- Een methode bindingsenergie die de bindingsenergie van het eiwit teruggeeft. Deze bindingsenergie moet berekend worden op basis van de vouwconfiguratie die ingesteld werd voor het eiwit. Indien er geen vouwconfiguratie werd ingesteld, dan moet 0 (nul) teruggegeven worden.
- Een methode __str__ waarmee een stringvoorstelling van het eiwit kan uitgeschreven worden volgens het formaat aangegeven in onderstaand voorbeeld. Hierbij moeten de ladingen van de aminozuren gecentreerd over vier posities weergegeven worden in de hokjes. Indien er een vouwconfiguratie werd ingesteld, dan moeten de schotjes op de juiste plaatsen weergegeven worden.
Voorbeeld
>>> eiwit = Eiwit([-2, 5, -4, 2, 4, -1, 2]) Traceback (most recent call last): AssertionError: ongeldige begintoestand >>> eiwit = Eiwit([-2, 5, -4, 2, 4, -1, 2, 1, 3, 2, -5, 2, 5, -3, 6, 1]) >>> eiwit.bindingsenergie() 0 >>> print(eiwit) +----+----+----+----+ | -2 5 -4 2 | + + + + + | 4 -1 2 1 | + + + + + | 3 2 -5 2 | + + + + + | 5 -3 6 1 | +----+----+----+----+ >>> eiwit.opvouwen([(1, 1, 1, 2), (2, 1, 2, 2), (3, 0, 3, 1), ... (1, 0, 2, 0), (0, 1, 1, 1), (1, 1, 2, 1), ... (0, 2, 1, 2), (2, 2, 3, 2), (1, 3, 2, 3)]) >>> eiwit.bindingsenergie() -58 >>> print(eiwit) +----+----+----+----+ | -2 5 -4 2 | + +----+----+ + | 4 -1 | 2 1 | +----+----+ +----+ | 3 2 | -5 2 | + + +----+ + | 5 | -3 6 1 | +----+----+----+----+ >>> eiwit.opvouwen([(1, 1, 1, 2), (2, 1, 2, 2), (4, 0, 4, 1)]) Traceback (most recent call last): AssertionError: ongeldige configuratie >>> eiwit.opvouwen([(1, 1, 1, 2), (2, 1, 3, 3), (3, 0, 3, 1)]) Traceback (most recent call last): AssertionError: ongeldige configuratie >>> eiwit = Eiwit([7, -2, -4, 3], [(0, 1, 1, 1)]) >>> eiwit.bindingsenergie() -6 >>> print(eiwit) +----+----+ | 7 -2 | + +----+ | -4 3 | +----+----+
| Added by: | Peter Dawyndt |
| Date: | 2012-08-13 |
| Time limit: | 5s-20s |
| Source limit: | 50000B |
| Memory limit: | 1536MB |
| Cluster: | Cube (Intel G860) |
| Languages: | PY_NBC |
| Resource: | None |

 RSS
RSS